Genvorhersage - Gene prediction

In Computational Biology , Genvorhersage oder Gen Feststellung bezieht sich auf den Prozess der Regionen genomischer DNA codieren , identifiziert Gene . Dies umfasst sowohl Protein-kodierende Gene als auch RNA-Gene , kann aber auch die Vorhersage anderer funktioneller Elemente wie regulatorischer Regionen umfassen . Das Auffinden von Genen ist einer der ersten und wichtigsten Schritte zum Verständnis des Genoms einer Art nach deren Sequenzierung .
In seinen frühesten Tagen beruhte "Genfindung" auf sorgfältigen Experimenten an lebenden Zellen und Organismen. Die statistische Analyse der Raten der homologen Rekombination mehrerer verschiedener Gene könnte ihre Reihenfolge auf einem bestimmten Chromosom bestimmen , und Informationen aus vielen solchen Experimenten könnten kombiniert werden, um eine genetische Karte zu erstellen , die die grobe Position bekannter Gene relativ zueinander spezifiziert. Mit einer umfassenden Genomsequenz und leistungsstarken Rechenressourcen, die der Forschungsgemeinschaft zur Verfügung stehen, wurde die Genfindung heute als weitgehend rechnerisches Problem neu definiert.
Die Bestimmung, ob eine Sequenz funktionsfähig ist, sollte von der Bestimmung der Funktion des Gens oder seines Produkts unterschieden werden. Die Vorhersage der Funktion eines Gens und die Bestätigung, dass die Genvorhersage genau ist, erfordert immer noch In-vivo- Experimente durch Gen-Knockout und andere Assays, obwohl die Grenzen der Bioinformatik- Forschung es zunehmend ermöglichen, die Funktion eines Gens allein anhand seiner Sequenz vorherzusagen.
Die Genvorhersage ist einer der Schlüsselschritte bei der Annotation des Genoms nach der Sequenzassemblierung , der Filterung nichtkodierender Regionen und der wiederholten Maskierung.
Die Genvorhersage steht in engem Zusammenhang mit dem sogenannten "Zielsuchproblem", bei dem untersucht wird, wie DNA-bindende Proteine ( Transkriptionsfaktoren ) bestimmte Bindungsstellen im Genom lokalisieren . Viele Aspekte der Strukturgen-Vorhersage basieren auf den gegenwärtigen Verständnis der zu Grunde liegenden biochemischen Vorgänge in der Zelle , wie Gen - Transkription , Translation , Protein-Protein - Wechselwirkungen und Regelungsprozesse , die in den verschiedenen Gegenstand aktiver Forschung sind omics Bereichen wie Transkriptomik , Proteomics , Metabolomics und allgemeiner strukturelle und funktionelle Genomics .
Empirische Methoden
In empirischen (Ähnlichkeits-, Homologie- oder evidenzbasierten) Genfindungssystemen wird das Zielgenom nach Sequenzen durchsucht, die extrinsischen Beweisen in Form der bekannten exprimierten Sequenz-Tags , Messenger-RNA (mRNA), Proteinprodukte und homologen oder ähnlich sind orthologe Sequenzen. Bei einer mRNA-Sequenz ist es trivial, eine einzigartige genomische DNA-Sequenz abzuleiten, von der sie transkribiert werden musste . Bei gegebener Proteinsequenz kann eine Familie möglicher codierender DNA-Sequenzen durch umgekehrte Translation des genetischen Codes abgeleitet werden . Sobald Kandidaten-DNA-Sequenzen bestimmt wurden, ist es ein relativ einfaches algorithmisches Problem, ein Zielgenom effizient nach vollständigen oder teilweisen und genauen oder ungenauen Übereinstimmungen zu durchsuchen. Bei einer gegebenen Sequenz suchen lokale Ausrichtungsalgorithmen wie BLAST , FASTA und Smith-Waterman nach Regionen mit Ähnlichkeit zwischen der Zielsequenz und möglichen Kandidatenübereinstimmungen. Übereinstimmungen können vollständig oder teilweise und genau oder ungenau sein. Der Erfolg dieses Ansatzes wird durch den Inhalt und die Genauigkeit der Sequenzdatenbank begrenzt.
Ein hohes Maß an Ähnlichkeit mit einer bekannten Messenger-RNA oder einem bekannten Proteinprodukt ist ein starker Beweis dafür, dass eine Region eines Zielgenoms ein Protein-kodierendes Gen ist. Um diesen Ansatz systemisch anzuwenden, ist jedoch eine umfassende Sequenzierung von mRNA- und Proteinprodukten erforderlich. Dies ist nicht nur teuer, sondern in komplexen Organismen wird zu einem bestimmten Zeitpunkt nur eine Teilmenge aller Gene im Genom des Organismus exprimiert, was bedeutet, dass extrinsische Beweise für viele Gene in keiner einzelnen Zellkultur leicht zugänglich sind. Um extrinsische Beweise für die meisten oder alle Gene in einem komplexen Organismus zu sammeln, müssen daher viele hundert oder tausend Zelltypen untersucht werden , was weitere Schwierigkeiten mit sich bringt. Beispielsweise können einige menschliche Gene nur während der Entwicklung als Embryo oder Fötus exprimiert werden, was aus ethischen Gründen schwierig zu untersuchen sein könnte.
Trotz dieser Schwierigkeiten wurden umfangreiche Transkript- und Proteinsequenzdatenbanken für menschliche und andere wichtige Modellorganismen in der Biologie wie Mäuse und Hefen erstellt. Beispielsweise enthält die RefSeq- Datenbank Transkript- und Proteinsequenzen von vielen verschiedenen Spezies, und das Ensembl- System ordnet diese Beweise umfassend menschlichen und mehreren anderen Genomen zu. Es ist jedoch wahrscheinlich, dass diese Datenbanken sowohl unvollständig sind als auch kleine, aber erhebliche Mengen fehlerhafter Daten enthalten.
Neue Hochdurchsatz- Transkriptomsequenzierungstechnologien wie RNA-Seq- und ChIP-Sequenzierung eröffnen Möglichkeiten, zusätzliche extrinsische Beweise in die Genvorhersage und -validierung einzubeziehen, und ermöglichen eine strukturreiche und genauere Alternative zu früheren Methoden zur Messung der Genexpression wie dem exprimierten Sequenz-Tag oder DNA-Microarray .
Zu den größten Herausforderungen bei der Genvorhersage gehören die Behandlung von Sequenzierungsfehlern in DNA-Rohdaten, die Abhängigkeit von der Qualität der Sequenzassemblierung , der Umgang mit kurzen Lesevorgängen, Frameshift-Mutationen , überlappende Gene und unvollständige Gene.
Bei Prokaryoten ist es wichtig, den horizontalen Gentransfer bei der Suche nach Gensequenzhomologie zu berücksichtigen . Ein weiterer wichtiger Faktor, der in aktuellen Tools zum Nachweis von Genen nicht ausreichend genutzt wird, ist die Existenz von Genclustern - Operons (funktionierende DNA- Einheiten, die einen Cluster von Genen unter der Kontrolle eines einzelnen Promotors enthalten ) sowohl in Prokaryoten als auch in Eukaryoten. Die meisten gängigen Gendetektoren behandeln jedes Gen isoliert, unabhängig von anderen, was biologisch nicht korrekt ist.
Ab-initio- Methoden
Die Ab-Initio-Genvorhersage ist eine intrinsische Methode, die auf dem Gengehalt und der Signaldetektion basiert. Aufgrund des inhärenten Aufwands und der Schwierigkeit, extrinsische Beweise für viele Gene zu erhalten, ist es auch notwendig, auf die Ab-initio -Genfindung zurückzugreifen, bei der die genomische DNA-Sequenz allein systematisch nach bestimmten verräterischen Anzeichen von Protein-kodierenden Genen durchsucht wird. Diese Zeichen können grob entweder als Signale , spezifische Sequenzen, die auf das Vorhandensein eines Gens in der Nähe hinweisen, oder als Inhalt statistischer Eigenschaften der Protein-kodierenden Sequenz selbst kategorisiert werden . Ab-initio -Genfindung könnte genauer als Genvorhersage charakterisiert werden , da im Allgemeinen extrinsische Beweise erforderlich sind, um endgültig festzustellen, dass ein mutmaßliches Gen funktionsfähig ist.
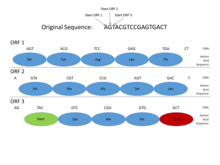
In den Genomen von Prokaryoten weisen Gene spezifische und relativ gut verstandene Promotorsequenzen (Signale) auf, wie die Pribnow-Box und Transkriptionsfaktor- Bindungsstellen , die leicht systematisch zu identifizieren sind. Die Sequenz, die für ein Protein kodiert, tritt auch als ein zusammenhängender offener Leserahmen (ORF) auf, der typischerweise viele hundert oder tausend Basenpaare lang ist. Die Statistiken der Stoppcodons sind so, dass selbst das Auffinden eines offenen Leserasters dieser Länge ein ziemlich informatives Zeichen ist. (Da 3 der 64 möglichen Codons im genetischen Code Stopcodons sind, würde man ungefähr alle 20–25 Codons oder 60–75 Basenpaare in einer zufälligen Sequenz ein Stopcodon erwarten .) Darüber hinaus hat die proteinkodierende DNA bestimmte Eigenschaften Periodizitäten und andere statistische Eigenschaften, die in einer Sequenz dieser Länge leicht zu erkennen sind. Diese Eigenschaften machen das Auffinden prokaryotischer Gene relativ einfach, und gut konzipierte Systeme können ein hohes Maß an Genauigkeit erreichen.
Die Ab-initio -Genfindung bei Eukaryoten , insbesondere bei komplexen Organismen wie Menschen, ist aus mehreren Gründen erheblich schwieriger. Erstens sind der Promotor und andere regulatorische Signale in diesen Genomen komplexer und weniger gut verstanden als in Prokaryoten, was es schwieriger macht, sie zuverlässig zu erkennen. Zwei klassische Beispiele für Signale, die von eukaryotischen Genfindern identifiziert wurden, sind CpG-Inseln und Bindungsstellen für einen Poly (A) -Schwanz .
Zweitens bedeuten Spleißmechanismen , die von eukaryotischen Zellen verwendet werden, dass eine bestimmte Protein-kodierende Sequenz im Genom in mehrere Teile ( Exons ) unterteilt ist, die durch nicht-kodierende Sequenzen ( Introns ) getrennt sind. (Spleißstellen sind selbst ein weiteres Signal, das eukaryotische Gensucher häufig identifizieren sollen.) Ein typisches Protein-kodierendes Gen beim Menschen kann in ein Dutzend Exons mit einer Länge von jeweils weniger als zweihundert Basenpaaren und einigen bis zu zwanzig Basenpaaren unterteilt werden bis dreißig. Es ist daher viel schwieriger, Periodizitäten und andere bekannte Inhaltseigenschaften von Protein-kodierender DNA in Eukaryoten nachzuweisen.
Fortgeschrittene Gensucher sowohl für prokaryotische als auch für eukaryotische Genome verwenden typischerweise komplexe probabilistische Modelle , wie beispielsweise Hidden-Markov-Modelle (HMMs), um Informationen aus einer Vielzahl verschiedener Signal- und Inhaltsmessungen zu kombinieren. Das GLIMMER- System ist ein weit verbreiteter und hochpräziser Gensucher für Prokaryoten. GeneMark ist ein weiterer beliebter Ansatz. Im Vergleich dazu haben eukaryotische Ab-initio -Gensucher nur begrenzten Erfolg erzielt; Bemerkenswerte Beispiele sind die GENSCAN- und Genid- Programme. Der SNAP-Gensucher basiert wie Genscan auf HMM und versucht, an verschiedene Organismen anpassungsfähiger zu werden, um Probleme im Zusammenhang mit der Verwendung eines Gensuchers für eine Genomsequenz zu lösen, gegen die er nicht trainiert wurde. Einige neuere Ansätze wie mSplicer, CONTRAST oder mGene verwenden auch Techniken des maschinellen Lernens wie Support-Vektor-Maschinen für eine erfolgreiche Genvorhersage. Sie erstellen ein Unterscheidungsmodell unter Verwendung versteckter Markov-Unterstützungsvektormaschinen oder bedingter Zufallsfelder , um eine genaue Funktion zur Bewertung der Genvorhersage zu erlernen.
Ab-Initio- Methoden wurden mit einer Empfindlichkeit von nahezu 100% verglichen. Mit zunehmender Empfindlichkeit leidet die Genauigkeit jedoch unter erhöhten Fehlalarmen .
Andere Signale
Unter den abgeleiteten Signalen für die Vorhersage verwendet werden , sind resultierende Statistiken aus den Subsequenz Statistiken wie k-mer - Statistiken, Isochore (Genetics) oder Zusammensetzungs Domäne GC Zusammensetzung / Uniformität / Entropie - Sequenz und Rahmenlänge, Intron / Exon / Donor / Acceptor / Promoter und Ribosomales Bindungsstellenvokabular , Fraktale Dimension , Fourier-Transformation einer Pseudo-Zahlen-codierten DNA, Z-Kurven- Parameter und bestimmte Laufmerkmale.
Es wurde vorgeschlagen, dass andere als die in Sequenzen direkt nachweisbaren Signale die Genvorhersage verbessern können. Beispielsweise wurde über die Rolle der Sekundärstruktur bei der Identifizierung von regulatorischen Motiven berichtet. Darüber hinaus wurde vorgeschlagen, dass die Vorhersage der RNA-Sekundärstruktur die Vorhersage der Spleißstelle unterstützt.
Neuronale Netze
Künstliche neuronale Netze sind Rechenmodelle, die sich durch maschinelles Lernen und Mustererkennung auszeichnen . Neuronale Netze müssen mit Beispieldaten trainiert werden, bevor experimentelle Daten verallgemeinert werden können, und anhand von Benchmarkdaten getestet werden. Neuronale Netze können ungefähre Lösungen für Probleme finden, die algorithmisch schwer zu lösen sind, sofern genügend Trainingsdaten vorhanden sind. Bei der Anwendung auf die Genvorhersage können neuronale Netze zusammen mit anderen Ab-initio- Methoden verwendet werden, um biologische Merkmale wie Spleißstellen vorherzusagen oder zu identifizieren. Ein Ansatz besteht darin, ein Schiebefenster zu verwenden, das die Sequenzdaten überlappend durchläuft. Die Ausgabe an jeder Position ist eine Bewertung, die darauf basiert, ob das Netzwerk glaubt, dass das Fenster eine Donor-Spleißstelle oder eine Akzeptor-Spleißstelle enthält. Größere Fenster bieten mehr Genauigkeit, erfordern aber auch mehr Rechenleistung. Ein neuronales Netzwerk ist ein Beispiel für einen Signalsensor, dessen Ziel es ist, eine funktionelle Stelle im Genom zu identifizieren.
Kombinierte Ansätze
Programme wie Maker kombinieren extrinsische und Ab-initio- Ansätze, indem sie Protein- und EST- Daten auf das Genom abbilden , um Ab-initio- Vorhersagen zu validieren . Augustus , der als Teil der Maker-Pipeline verwendet werden kann, kann auch Hinweise in Form von EST-Alignments oder Proteinprofilen enthalten, um die Genauigkeit der Genvorhersage zu erhöhen.
Vergleichende genomische Ansätze
Da das gesamte Genom vieler verschiedener Arten sequenziert wird, ist eine vielversprechende Richtung in der aktuellen Forschung zur Genfindung ein vergleichender genomischer Ansatz.
Dies basiert auf dem Prinzip, dass die Kräfte der natürlichen Selektion dazu führen, dass Gene und andere funktionelle Elemente langsamer mutieren als der Rest des Genoms, da Mutationen in funktionellen Elementen den Organismus eher negativ beeinflussen als Mutationen an anderer Stelle. Gene können somit durch Vergleichen der Genome verwandter Arten nachgewiesen werden, um diesen evolutionären Konservierungsdruck festzustellen. Dieser Ansatz wurde zuerst auf das Genom von Mäusen und Menschen angewendet, wobei Programme wie SLAM, SGP und TWINSCAN / N-SCAN und CONTRAST verwendet wurden.
Mehrere Informanten
TWINSCAN untersuchte nur die Syntenie zwischen Mensch und Maus, um nach orthologen Genen zu suchen. Programme wie N-SCAN und CONTRAST ermöglichten den Einbau von Alignments mehrerer Organismen oder im Fall von N-SCAN eines einzelnen alternativen Organismus vom Ziel. Die Verwendung mehrerer Informanten kann zu erheblichen Verbesserungen der Genauigkeit führen.
CONTRAST besteht aus zwei Elementen. Der erste ist ein kleinerer Klassifikator, der Donor-Spleißstellen und Akzeptor-Spleißstellen sowie Start- und Stoppcodons identifiziert. Das zweite Element beinhaltet die Erstellung eines vollständigen Modells mithilfe von maschinellem Lernen. Die Aufteilung des Problems in zwei Teile bedeutet, dass kleinere Zieldatensätze zum Trainieren der Klassifizierer verwendet werden können und dass der Klassifizierer unabhängig arbeiten und mit kleineren Fenstern trainiert werden kann. Das vollständige Modell kann den unabhängigen Klassifizierer verwenden und muss keine Rechenzeit oder Modellkomplexität verschwenden, um die Intron-Exon-Grenzen neu zu klassifizieren. In dem Artikel, in dem CONTRAST vorgestellt wird, wird vorgeschlagen, ihre Methode (und die von TWINSCAN usw.) als De-novo- Genassemblierung zu klassifizieren , indem alternative Genome verwendet und als von ab initio verschieden identifiziert werden , bei dem ein Ziel-Informantengenom verwendet wird.
Der vergleichende Genbefund kann auch verwendet werden, um qualitativ hochwertige Annotationen von einem Genom zum anderen zu projizieren. Bemerkenswerte Beispiele sind Projector, GeneWise, GeneMapper und GeMoMa. Solche Techniken spielen jetzt eine zentrale Rolle bei der Annotation aller Genome.
Pseudogene Vorhersage
Pseudogene sind nahe Verwandte von Genen, die eine sehr hohe Sequenzhomologie aufweisen, jedoch nicht in der Lage sind, für dasselbe Proteinprodukt zu kodieren . Während sie einst als Nebenprodukte der Gensequenzierung verbannt wurden , werden sie zunehmend zu eigenständigen prädiktiven Zielen, da regulatorische Rollen aufgedeckt werden. Die Pseudogenvorhersage nutzt vorhandene Sequenzähnlichkeits- und Ab-initio-Methoden und fügt zusätzliche Filter und Methoden zur Identifizierung von Pseudogenmerkmalen hinzu.
Sequenzähnlichkeitsmethoden können für die Pseudogenvorhersage unter Verwendung zusätzlicher Filterung angepasst werden, um mögliche Pseudogene zu finden. Dies könnte die Deaktivierungserkennung verwenden, die nach Unsinn- oder Frameshift-Mutationen sucht, die eine ansonsten funktionale Codierungssequenz abschneiden oder kollabieren würden. Darüber hinaus kann die Translation von DNA in Proteinsequenzen effektiver sein als nur eine reine DNA-Homologie.
Inhaltssensoren können nach den Unterschieden in den statistischen Eigenschaften zwischen Pseudogenen und Genen gefiltert werden, z. B. nach einer verringerten Anzahl von CpG-Inseln in Pseudogenen oder nach Unterschieden im GC-Gehalt zwischen Pseudogenen und ihren Nachbarn. Signalsensoren können auch auf Pseudogene geschliffen werden, um das Fehlen von Introns oder Polyadeninschwänzen festzustellen.
Metagenomische Genvorhersage
Metagenomik ist die Untersuchung von genetischem Material, das aus der Umwelt gewonnen wird und zu Sequenzinformationen aus einem Pool von Organismen führt. Die Vorhersage von Genen ist nützlich für die vergleichende Metagenomik .
Metagenomics-Tools fallen auch in die Grundkategorien der Verwendung von Sequenzähnlichkeitsansätzen (MEGAN4) und Ab-initio-Techniken (GLIMMER-MG).
Glimmer-MG ist eine Erweiterung von GLIMMER , die sich hauptsächlich auf einen Ab-initio-Ansatz zur Genfindung und zur Verwendung von Trainingssätzen verwandter Organismen stützt. Die Vorhersagestrategie wird durch die Klassifizierung und Clusterbildung von Gendatensätzen vor der Anwendung von Ab-initio-Genvorhersagemethoden ergänzt. Die Daten sind nach Arten gruppiert. Diese Klassifizierungsmethode nutzt Techniken aus der metagenomischen phylogenetischen Klassifizierung. Ein Beispiel für Software für diesen Zweck ist Phymm, das interpolierte Markov-Modelle verwendet, und PhymmBL, das BLAST in die Klassifizierungsroutinen integriert.
MEGAN4 verwendet einen Sequenzähnlichkeitsansatz, bei dem die lokale Ausrichtung mit Datenbanken bekannter Sequenzen verwendet wird, versucht jedoch auch, anhand zusätzlicher Informationen zu funktionellen Rollen, biologischen Pfaden und Enzymen zu klassifizieren. Wie bei der Vorhersage von Genen einzelner Organismen sind Sequenzähnlichkeitsansätze durch die Größe der Datenbank begrenzt.
FragGeneScan und MetaGeneAnnotator sind beliebte Genvorhersageprogramme, die auf dem Hidden Markov-Modell basieren . Diese Prädiktoren berücksichtigen Sequenzierungsfehler, Teilgene und arbeiten für kurze Lesevorgänge.
Ein weiteres schnelles und genaues Werkzeug zur Genvorhersage in Metagenomen ist MetaGeneMark. Dieses Tool wird vom DOE Joint Genome Institute verwendet, um IMG / M, die bislang größte Metagenomsammlung, mit Anmerkungen zu versehen.
Siehe auch
- Liste der Gen-Vorhersage-Software
- Sequence Mining
- Vorhersage der Proteinfunktion
- Phylogenetischer Fußabdruck
- Sequenzähnlichkeit (Homologie)
Externe Links
- Augustus
- FGENESH
- GeMoMa - Homologiebasierte Genvorhersage basierend auf der Erhaltung der Aminosäure- und Intronposition sowie RNA-Seq-Daten
- Genid , SGP2
- Glimmer , GlimmerHMM
- GenomeThreader
- ChemGenome
- GeneMark
- Gismo
- mGene
- StarORF - Ein plattformübergreifendes und Web-Tool zur Vorhersage von ORFs und zur Ermittlung der umgekehrten Komplementsequenz
- Maker - Eine tragbare und einfach zu konfigurierende Pipeline für Genomanmerkungen
