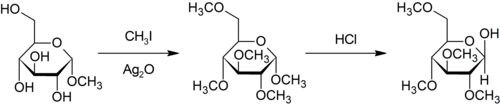Methylierung - Methylation
In den chemischen Wissenschaften bezeichnet Methylierung die Addition einer Methylgruppe an ein Substrat oder die Substitution eines Atoms (oder einer Gruppe) durch eine Methylgruppe. Methylierung ist eine Form der Alkylierung , bei der eine Methylgruppe ein Wasserstoffatom ersetzt. Diese Begriffe werden häufig in der Chemie , Biochemie , Bodenkunde und den biologischen Wissenschaften verwendet .
In biologischen Systemen wird die Methylierung durch Enzyme katalysiert ; eine solche Methylierung kann an der Modifikation von Schwermetallen , der Regulation der Genexpression , der Regulation der Proteinfunktion und der RNA-Prozessierung beteiligt sein . Die In-vitro-Methylierung von Gewebeproben ist auch eine Methode zur Reduzierung bestimmter histologischer Färbeartefakte . Das Gegenstück zur Methylierung heißt Demethylierung .
In Biologie
In biologischen Systemen erfolgt die Methylierung durch Enzyme. Methylierung kann Schwermetalle modifizieren, Genexpression, RNA-Verarbeitung und Proteinfunktion regulieren. Es wurde als ein Schlüsselprozess erkannt, der der Epigenetik zugrunde liegt .
Methanogenese
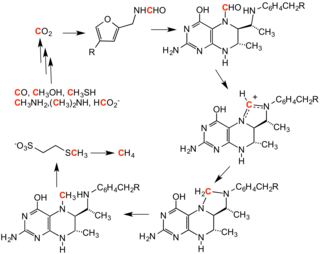
Die Methanogenese , der Prozess, bei dem Methan aus CO 2 erzeugt wird , umfasst eine Reihe von Methylierungsreaktionen. Diese Reaktionen werden durch eine Reihe von Enzymen bewirkt, die von einer Familie anaerober Mikroben beherbergt werden.
Bei der reversen Methanogenese dient Methan als Methylierungsmittel.
O-Methyltransferasen
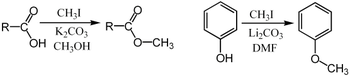
Eine Vielzahl von Phenolen unterliegt einer O-Methylierung, um Anisolderivate zu ergeben . Dieses Verfahren, katalysiert durch Enzyme wie Caffeoyl-CoA - O-methyltransferase , ist eine Schlüsselreaktion in der Biosynthese von lignols , Vorläufern zu Lignin , einem Hauptstrukturkomponente von Pflanzen.
Pflanzen produzieren Flavonoide und Isoflavone mit Methylierungen an Hydroxylgruppen, dh Methoxybindungen . Diese 5-O-Methylierung beeinflusst die Wasserlöslichkeit der Flavonoide. Beispiele sind 5-O-Methylgenistein , 5-O-Methylmyricetin oder 5-O-Methylquercetin , auch bekannt als Azaleatin.
Proteine
Zusammen mit Ubiquitin und Phosphorylierung ist die Methylierung ein wichtiger biochemischer Prozess zur Modifizierung der Proteinfunktion. Die häufigsten Proteinmethylierungen produzieren spezifische Histone aus Arginin und Lysin. Ansonsten sind Histidin, Glutamat, Asparagin, Cystein anfällig für Methylierung. Einige dieser Produkte umfassen S- Methylcystein , zwei Isomere von N- Methylhistidin und zwei Isomere von N- Methylarginin.
Methioninsynthase

Methioninsynthase regeneriert Methionin (Met) aus Homocystein (Hcy). Die Gesamtreaktion wandelt 5-Methyltetrahydrofolat (N 5 -MeTHF) in Tetrahydrofolat (THF) um, während eine Methylgruppe auf Hcy übertragen wird, um Met zu bilden. Methionin-Synthasen können Cobalamin-abhängig und Cobalamin-unabhängig sein: Pflanzen haben beides, Tiere sind von der Methylcobalamin-abhängigen Form abhängig.
Bei Methylcobalamin-abhängigen Formen des Enzyms verläuft die Reaktion in einer Ping-Pong-Reaktion in zwei Schritten. Das Enzym wird zunächst durch die Übertragung einer Methylgruppe von N 5 -MeTHF auf Co(I) in enzymgebundenem Cobalamin (Cob) in einen reaktiven Zustand versetzt , wodurch Methyl-Cobalamin (Me-Cob) gebildet wird, das nun Me-Co . enthält (III) und Aktivierung des Enzyms. Dann reagiert ein Hcy, das an ein enzymgebundenes Zink koordiniert hat , um ein reaktives Thiolat zu bilden, mit dem Me-Cob. Die aktivierte Methylgruppe wird von Me-Cob auf das Hcy-Thiolat übertragen, das Co(I) in Cob regeneriert, und Met wird aus dem Enzym freigesetzt.
Schwermetalle: Arsen, Quecksilber, Cadmium
Biomethylierung ist der Weg zur Umwandlung einiger schwerer Elemente in beweglichere oder tödlichere Derivate, die in die Nahrungskette gelangen können . Die Biomethylierung von Arsenverbindungen beginnt mit der Bildung von Methanarsonaten . So werden dreiwertige anorganische Arsenverbindungen zu Methanarsonat methyliert. S-Adenosylmethionin ist der Methyldonor. Die Methanarsonate sind die Vorläufer von Dimethylarsonaten, wiederum durch den Reduktionszyklus (zur Methylarsonsäure) gefolgt von einer zweiten Methylierung. Ähnliche Wege gelten für die Biosynthese von Methylquecksilber .
Epigenetische Methylierung
DNA/RNA-Methylierung
DNA-Methylierung bei Vertebraten erfolgt typischerweise an CpG-Stellen (Cytosin-Phosphat-Guanin-Stellen – d. h. dort, wo einem Cytosin direkt ein Guanin in der DNA-Sequenz folgt ). Diese Methylierung führt zur Umwandlung des Cytosins in 5-Methylcytosin . Die Bildung von Me-CpG wird durch das Enzym DNA-Methyltransferase katalysiert . Bei Säugetieren ist die DNA-Methylierung in Körperzellen üblich, und die Methylierung von CpG-Stellen scheint der Standard zu sein. In der menschlichen DNA sind etwa 80–90% der CpG-Stellen methyliert, aber es gibt bestimmte Bereiche, die als CpG-Inseln bekannt sind, die CG-reich sind (hoher Cytosin- und Guaningehalt, bestehend aus etwa 65% CG- Resten ), wobei keine methyliert ist . Diese sind mit den Promotoren von 56% der Säugetiergene verbunden, einschließlich aller ubiquitär exprimierten Gene . Ein bis zwei Prozent des menschlichen Genoms sind CpG-Cluster, und es besteht eine umgekehrte Beziehung zwischen CpG-Methylierung und Transkriptionsaktivität. Methylierung, die zur epigenetischen Vererbung beiträgt, kann entweder durch DNA-Methylierung oder Proteinmethylierung erfolgen. Unsachgemäße Methylierungen menschlicher Gene können zur Entwicklung von Krankheiten, einschließlich Krebs, führen. In ähnlicher Weise tritt RNA-Methylierung in verschiedenen RNA-Spezies auf, nämlich. tRNA , rRNA , mRNA , tmRNA , snRNA , snoRNA , miRNA und virale RNA. Für die RNA-Methylierung durch eine Vielzahl von RNA-Methyltransferasen werden verschiedene katalytische Strategien verwendet. Es wird angenommen, dass die RNA-Methylierung vor der DNA-Methylierung in den frühen Lebensformen auf der Erde existiert hat.
N6-Methyladenosin (m6A) ist die häufigste und am häufigsten vorkommende Methylierungsmodifikation in RNA-Molekülen (mRNA), die in Eukaryoten vorkommen. 5-Methylcytosin (5-mC) kommt auch häufig in verschiedenen RNA-Molekülen vor. Jüngste Daten deuten stark darauf hin, dass die m6A- und 5-mC-RNA-Methylierung die Regulation verschiedener biologischer Prozesse wie RNA-Stabilität und mRNA-Translation beeinflusst und dass eine abnorme RNA-Methylierung zur Ätiologie menschlicher Krankheiten beiträgt.
Proteinmethylierung
Die Proteinmethylierung findet typischerweise an Arginin- oder Lysin- Aminosäureresten in der Proteinsequenz statt. Arginin kann einmal (monomethyliertes Arginin) oder zweimal methyliert werden, entweder mit beiden Methylgruppen an einem terminalen Stickstoff ( asymmetrisches Dimethylarginin ) oder einer an beiden Stickstoffen (symmetrisches Dimethylarginin), durch Protein-Arginin-Methyltransferasen (PRMTs). Lysin kann einmal, zweimal oder dreimal durch Lysin-Methyltransferasen methyliert werden . Die Proteinmethylierung wurde in den Histonen am besten untersucht . Die Übertragung von Methylgruppen von S-Adenosylmethionin auf Histone wird durch Enzyme katalysiert, die als Histon-Methyltransferasen bekannt sind . Histone, die an bestimmten Resten methyliert sind, können epigenetisch wirken , um die Genexpression zu unterdrücken oder zu aktivieren. Proteinmethylierung ist eine Art posttranslationaler Modifikation .
Evolution
Der Methylmetabolismus ist sehr alt und findet sich in allen Organismen auf der Erde, von Bakterien bis zum Menschen, was auf die Bedeutung des Methylmetabolismus für die Physiologie hinweist. Tatsächlich verursacht die pharmakologische Hemmung der globalen Methylierung in Spezies wie Mensch, Maus, Fisch, Fliege, Fadenwurm, Pflanze, Alge und Cyanobakterien die gleichen Auswirkungen auf ihre biologischen Rhythmen, was die konservierte physiologische Rolle der Methylierung während der Evolution demonstriert.
In Chemie
Der Begriff Methylierung bezieht sich in der organischen Chemie auf den Alkylierungsprozess , der verwendet wird, um die Abgabe einer CH 3 -Gruppe zu beschreiben .
Elektrophile Methylierung
Methylierungen werden üblicherweise unter Verwendung elektrophiler Methylquellen wie Jodmethan , Dimethylsulfat , Dimethylcarbonat oder Tetramethylammoniumchlorid durchgeführt . Weniger verbreitete, aber stärkere (und gefährlichere) Methylierungsreagenzien sind Methyltriflat , Diazomethan und Methylfluorsulfonat ( magisches Methyl ). Diese Reagenzien reagieren alle über nukleophile S N 2 -Substitutionen . Zum Beispiel kann ein Carboxylat kann an Sauerstoff methyliert werden , um einen zur Erzielung von Methyl - Ester ; ein Alkoxidsalz RO – kann ebenfalls zu einem Ether , ROCH 3 methyliert werden ; oder ein Ketonenolat kann an Kohlenstoff methyliert werden, um ein neues Keton zu erzeugen .
Die Purdie-Methylierung ist eine Spezifische für die Methylierung an Sauerstoff von Kohlenhydraten unter Verwendung von Jodmethan und Silberoxid .
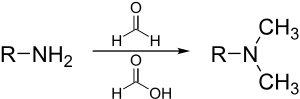
Eschweiler-Clarke-Methylierung
Die Eschweiler-Clarke-Reaktion ist eine Methode zur Methylierung von Aminen . Dieses Verfahren vermeidet das Risiko einer Quaternisierung , die auftritt, wenn Amine mit Methylhalogeniden methyliert werden.
Diazomethan und Trimethylsilyldiazomethan
Diazomethan und das sicherere Analogon Trimethylsilyldiazomethanmethylat Carbonsäuren, Phenole und sogar Alkohole:
- RCO 2 H + tmsCHN 2 + CH 3 OH → RCO 2 CH 3 + CH 3 Otms + N 2
Das Verfahren bietet den Vorteil, dass die Nebenprodukte leicht aus dem Produktgemisch entfernt werden.
Nucleophile Methylierung
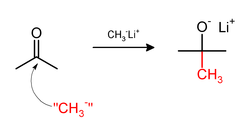
Bei der Methylierung werden manchmal nukleophile Methylreagenzien verwendet. Stark nukleophile Methylierungsmittel umfassen Methyllithium (CH 3 Li) oder Grignard-Reagenzien wie Methylmagnesiumbromid (CH 3 MgX). CH 3 Li fügt beispielsweise dem Carbonyl ( C=O ) von Ketonen und Aldehyden Methylgruppen hinzu .:
Mildere Methylierungsmittel umfassen Tetramethylzinn , Dimethylzink und Trimethylaluminium .
Siehe auch
Biologie-Themen
- Bisulfit-Sequenzierung – die biochemische Methode zur Bestimmung des Vorhandenseins oder Fehlens von Methylgruppen in einer DNA-Sequenz
- MethDB DNA-Methylierungsdatenbank
- Mikroskalige Thermophorese – eine biophysikalische Methode zur Bestimmung des Methylierungszustandes von DNA
Themen der Organischen Chemie
- Alkylierung
- Methoxy
- Titan-Zink-Methylenierung
- Petasis-Reagenz
- Nysted-Reagenz
- Wittig-Reaktion
- Tebbes Reagenz
Verweise
Externe Links
- deltaMasses Nachweis von Methylierungen nach Massenspektrometrie