Zwei-Hybrid-Screening - Two-hybrid screening
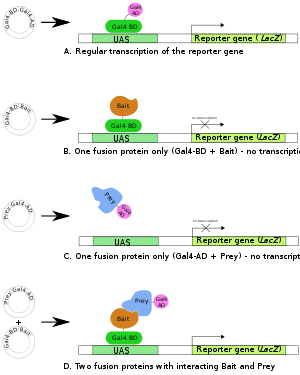
Ein . Das Gal4- Transkriptionsfaktor-Gen produziert ein Zwei-Domänen-Protein ( BD und AD ), das für die Transkription des Reportergens ( LacZ ) essentiell ist .
B , C . Zwei Fusionsproteine werden hergestellt: Gal4BD+Bait und Gal4AD+Prey . Keines von ihnen reicht normalerweise allein aus, um die Transkription (des Reportergens) zu initiieren.
D . Wenn beide Fusionsproteine produziert werden und der Bait-Teil des ersten Fusionsproteins mit dem Prey-Teil des zweiten interagiert, erfolgt die Transkription des Reportergens.
Das Zwei-Hybrid-Screening (ursprünglich als Hefe-Zwei-Hybrid-System oder Y2H bekannt ) ist eine molekularbiologische Technik, die verwendet wird, um Protein-Protein-Wechselwirkungen (PPIs) und Protein-DNA-Wechselwirkungen zu entdecken, indem auf physikalische Wechselwirkungen (wie Bindung) zwischen zwei Proteinen getestet wird oder ein einzelnes Protein bzw. ein DNA- Molekül.
Die Prämisse hinter dem Test ist die Aktivierung von Downstream- Reportergen (en) durch die Bindung eines Transkriptionsfaktors an eine Upstream-Aktivierungssequenz (UAS). Beim Two-Hybrid-Screening wird der Transkriptionsfaktor in zwei separate Fragmente aufgespalten, die DNA-bindende Domäne (DBD oder oft auch als BD abgekürzt) und aktivierende Domäne (AD). Die BD ist die Domäne, die für die Bindung an die UAS verantwortlich ist, und die AD ist die Domäne, die für die Aktivierung der Transkription verantwortlich ist . Der Y2H ist somit ein Proteinfragment-Komplementationsassay .
Geschichte
Die 1989 von Stanley Fields und Ok-Kyu Song entwickelte Technik wurde ursprünglich entwickelt, um Protein-Protein-Wechselwirkungen mit dem Transkriptionsaktivator Gal4 der Hefe Saccharomyces cerevisiae nachzuweisen . Das Gal4- Protein aktivierte die Transkription eines Gens, das an der Galactose-Verwertung beteiligt ist und die Grundlage der Selektion bildete. Seitdem wurde das gleiche Prinzip angepasst, um viele alternative Methoden zu beschreiben, darunter einige, die Protein-DNA-Wechselwirkungen oder DNA-DNA-Wechselwirkungen nachweisen , sowie Methoden, die verschiedene Wirtsorganismen wie Escherichia coli oder Säugerzellen anstelle von Hefe verwenden.
Grundvoraussetzung
Der Schlüssel zum Zwei-Hybrid-Screening besteht darin, dass bei den meisten eukaryotischen Transkriptionsfaktoren die aktivierenden und bindenden Domänen modular sind und ohne direkte Bindung in unmittelbarer Nähe zueinander funktionieren können. Dies bedeutet, dass der Transkriptionsfaktor, obwohl er in zwei Fragmente gespalten ist, immer noch die Transkription aktivieren kann, wenn die beiden Fragmente indirekt verbunden sind.
Der gebräuchlichste Screening-Ansatz ist der Hefe-Zwei-Hybrid-Assay. Bei diesem Ansatz weiß der Forscher, wo sich jede Beute auf dem verwendeten Medium (Agarplatten) befindet. Millionen potenzieller Interaktionen in mehreren Organismen wurden im letzten Jahrzehnt mithilfe von Hochdurchsatz-Screening- Systemen (oft unter Verwendung von Robotern) gescreent und über Tausende von Interaktionen wurden erkannt und in Datenbanken als BioGRID kategorisiert . Dieses System verwendet oft einen gentechnisch veränderten Hefestamm, bei dem die Biosynthese bestimmter Nährstoffe (normalerweise Aminosäuren oder Nukleinsäuren ) fehlt. Wenn sie auf Medien angebaut werden, denen diese Nährstoffe fehlen, überlebt die Hefe nicht. Dieser mutierte Hefestamm kann dazu gebracht werden, fremde DNA in Form von Plasmiden einzubauen . Beim Hefe-Zwei-Hybrid-Screening werden separate Köder- und Beute-Plasmide gleichzeitig in den mutierten Hefestamm eingeführt, oder es wird eine Paarungsstrategie verwendet, um beide Plasmide in eine Wirtszelle zu bringen.
Der zweite Hochdurchsatz-Ansatz ist der Bibliotheks-Screening-Ansatz. Bei dieser Anordnung werden die Köder- und Beutezellen in zufälliger Reihenfolge gepaart. Nach der Paarung und Selektion überlebender Zellen auf selektivem Medium wird der Wissenschaftler die isolierten Plasmide sequenzieren, um zu sehen, welche Beute (DNA-Sequenz) mit dem verwendeten Köder interagiert. Dieser Ansatz weist eine niedrigere Reproduzierbarkeitsrate auf und führt im Vergleich zum Matrixansatz tendenziell zu höheren Mengen an falsch positiven Ergebnissen.
Plasmide werden manipuliert, um ein Proteinprodukt zu produzieren, in dem das DNA-bindende Domänen-(BD)-Fragment an ein Protein fusioniert ist, während ein anderes Plasmid manipuliert wird, um ein Proteinprodukt zu produzieren, in dem das Aktivierungsdomänen-(AD)-Fragment an ein anderes Protein fusioniert ist. Das an die BD fusionierte Protein kann als Köderprotein bezeichnet werden und ist typischerweise ein bekanntes Protein, das der Forscher verwendet, um neue Bindungspartner zu identifizieren. Das an AD fusionierte Protein kann als Beuteprotein bezeichnet werden und kann entweder ein einzelnes bekanntes Protein oder eine Bibliothek bekannter oder unbekannter Proteine sein. In diesem Zusammenhang kann eine Bibliothek aus einer Sammlung von Protein-kodierenden Sequenzen bestehen, die alle Proteine darstellen, die in einem bestimmten Organismus oder Gewebe exprimiert werden, oder kann durch Synthese zufälliger DNA-Sequenzen erzeugt werden. Unabhängig von der Quelle werden sie anschließend in die proteinkodierende Sequenz eines Plasmids eingebaut, das dann in die für das Screening-Verfahren ausgewählten Zellen transfiziert wird. Diese Technik geht bei Verwendung einer Bibliothek davon aus, dass jede Zelle mit nicht mehr als einem einzelnen Plasmid transfiziert wird und dass daher jede Zelle letztendlich nicht mehr als ein einzelnes Mitglied der Proteinbibliothek exprimiert.
Wenn die Köder- und Beuteproteine interagieren (dh binden), dann sind AD und BD des Transkriptionsfaktors indirekt verbunden, wodurch die AD in die Nähe der Transkriptionsstartstelle gebracht wird und die Transkription von Reportergen(en) erfolgen kann. Wenn die beiden Proteine nicht interagieren, findet keine Transkription des Reportergens statt. Auf diese Weise ist eine erfolgreiche Interaktion zwischen dem Fusionsprotein mit einer Veränderung des Zellphänotyps verbunden.
Die Herausforderung, Zellen, die Proteine exprimieren, die zufällig mit ihren Gegenstück-Fusionsproteinen interagieren, von solchen zu trennen, die dies nicht tun, wird im folgenden Abschnitt behandelt.
Feste Domänen
In jeder Studie werden einige der zu untersuchenden Proteindomänen entsprechend den Zielen der Studie variiert, während andere Domänen, die selbst nicht untersucht werden, konstant gehalten werden. Beispielsweise wird in einer Zwei-Hybrid-Studie zur Auswahl von DNA-bindenden Domänen die DNA-bindende Domäne BD variiert, während die beiden interagierenden Proteine, der Köder und die Beute, konstant gehalten werden müssen, um eine starke Bindung zwischen den BD . aufrechtzuerhalten und AD. Es gibt eine Reihe von Domänen, aus denen BD, Köder und Beute und AD ausgewählt werden können, wenn diese konstant bleiben sollen. In Protein-Protein-Interaktionsuntersuchungen kann die BD aus einer von vielen starken DNA-bindenden Domänen wie Zif268 ausgewählt werden . Eine häufige Wahl von Köder- und Beutedomänen sind die Reste 263–352 von Hefe Gal11P mit einer N342V-Mutation bzw. die Reste 58–97 von Hefe Gal4. Diese Domänen können sowohl in hefe- als auch bakterienbasierten Selektionstechniken verwendet werden und sind dafür bekannt, stark aneinander zu binden.
Die gewählte AD muss in der Lage sein, die Transkription des Reportergens unter Verwendung der zelleigenen Transkriptionsmaschinerie zu aktivieren. Somit ist die Vielfalt von ADs, die zur Verwendung in hefebasierten Techniken verfügbar sind, möglicherweise nicht für die Verwendung in ihren auf Bakterien basierenden Analoga geeignet. Die vom Herpes-simplex-Virus abgeleiteten AD, VP16 und Hefe Gal4 AD wurden mit Erfolg in Hefe verwendet, während ein Teil der &agr;-Untereinheit der E. coli- RNA-Polymerase in E. coli- basierten Verfahren verwendet wurde.
Während stark aktivierende Domänen eine größere Empfindlichkeit gegenüber schwächeren Wechselwirkungen ermöglichen können, kann umgekehrt eine schwächere AD eine größere Stringenz bereitstellen.
Konstruktion von Expressionsplasmiden
Eine Anzahl von gentechnisch veränderten genetischen Sequenzen muss in die Wirtszelle eingebaut werden, um eine Zwei-Hybrid-Analyse oder eine ihrer abgeleiteten Techniken durchzuführen. Die Überlegungen und Methoden, die bei der Konstruktion und Abgabe dieser Sequenzen verwendet werden, unterscheiden sich je nach den Anforderungen des Assays und dem als experimenteller Hintergrund gewählten Organismus.
Es gibt zwei breite Kategorien von Hybridbibliotheken: Zufallsbibliotheken und cDNA-basierte Bibliotheken. Eine cDNA-Bibliothek wird durch die cDNA gebildet, die durch reverse Transkription von mRNA produziert wird, die aus spezifischen Zellen von Zelltypen gesammelt wurde. Diese Bibliothek kann in ein Konstrukt ligiert werden, so dass sie an die im Assay verwendete BD oder AD angehängt wird. Eine Zufallsbibliothek verwendet DNA-Längen mit Zufallssequenz anstelle dieser cDNA-Abschnitte. Zur Herstellung dieser Zufallssequenzen existiert eine Reihe von Verfahren, einschließlich der Kassettenmutagenese . Unabhängig von der Quelle der DNA-Bibliothek wird diese unter Verwendung der entsprechenden Restriktionsendonukleasen an die entsprechende Stelle im relevanten Plasmid/Phagemid ligiert .
E. coli -spezifische Überlegungen
Indem die Hybridproteine unter die Kontrolle von IPTG- induzierbaren lac- Promotoren gestellt werden , werden sie nur auf mit IPTG ergänzten Medien exprimiert. Durch Einschließen unterschiedlicher Antibiotikaresistenzgene in jedes genetische Konstrukt wird das Wachstum von nicht-transformierten Zellen durch Kultur auf Medien, die die entsprechenden Antibiotika enthalten, leicht verhindert. Dies ist besonders wichtig für Gegenselektionsverfahren, bei denen eine fehlende Interaktion für das Zellüberleben erforderlich ist.
Das Reportergen kann in das Genom von E. coli eingefügt werden, indem es zuerst in ein Episom eingefügt wird , eine Art von Plasmid mit der Fähigkeit, sich mit einer Kopienzahl von ungefähr einer pro Zelle selbst in das Genom der Bakterienzelle einzubauen.
Die Hybridexpressions-Phagemide können in E. coli XL-1 Blue-Zellen elektroporiert werden, die nach Amplifikation und Infektion mit VCS-M13- Helferphagen einen Vorrat an Bibliotheksphagen ergeben. Diese Phagen enthalten jeweils ein einzelsträngiges Mitglied der Phagemid-Bibliothek.
Wiederherstellung von Proteininformationen
Nach erfolgter Selektion muss die Primärstruktur der Proteine, die die entsprechenden Eigenschaften aufweisen, bestimmt werden. Dies wird durch das Abrufen der Protein-kodierenden Sequenzen (wie ursprünglich eingefügt) aus den Zellen erreicht, die den entsprechenden Phänotyp zeigen.
E coli
Das zur Transformation von E. coli- Zellen verwendete Phagemid kann aus den ausgewählten Zellen "gerettet" werden, indem sie mit VCS-M13-Helferphagen infiziert werden. Die resultierenden Phagenpartikel, die produziert werden, enthalten die einzelsträngigen Phagemide und werden verwendet, um XL-1 Blue-Zellen zu infizieren. Die doppelsträngigen Phagemide werden anschließend von diesen XL-1 Blue-Zellen gesammelt, was im Wesentlichen das Verfahren umkehrt, das verwendet wurde, um die ursprünglichen Phagen der Bibliothek zu produzieren. Schließlich werden die DNA-Sequenzen durch Didesoxy-Sequenzierung bestimmt .
Steuerung der Empfindlichkeit
Der von Escherichia coli abgeleitete Tet-R- Repressor kann analog zu einem konventionellen Reportergen verwendet und durch Tetracyclin oder Doxicyclin (Tet-R-Inhibitoren) kontrolliert werden. Somit wird die Expression von Tet-R durch das Standard-Zwei-Hybrid-System kontrolliert, aber das Tet-R kontrolliert (reprimiert) wiederum die Expression eines zuvor erwähnten Reporters wie HIS3 durch seinen Tet-R-Promotor. Tetracyclin oder seine Derivate können dann verwendet werden, um die Empfindlichkeit eines Systems unter Verwendung von Tet-R zu regulieren.
Die Empfindlichkeit kann auch durch Variieren der Abhängigkeit der Zellen von ihren Reportergenen gesteuert werden. Dies kann beispielsweise beeinflusst werden, indem die Konzentration von Histidin im Wachstumsmedium für his3- abhängige Zellen und die Konzentration von Streptomycin für aadA- abhängige Zellen verändert wird. Die Selektionsgen-Abhängigkeit kann auch durch Anwendung eines Inhibitors des Selektionsgens in einer geeigneten Konzentration kontrolliert werden. 3-Amino-1,2,4-triazol (3-AT) zum Beispiel ist ein kompetitiver Inhibitor des HIS3- Genprodukts und kann verwendet werden, um das minimale Niveau der HIS3- Expression zu titrieren, das für das Wachstum auf Histidin-defizienten Medien erforderlich ist.
Die Empfindlichkeit kann auch durch Variieren der Anzahl der Operatorsequenzen in der Reporter-DNA moduliert werden.
Nicht-Fusionsproteine
Ein drittes Nicht-Fusionsprotein kann mit zwei Fusionsproteinen coexprimiert werden. Je nach Untersuchung kann das dritte Protein eines der Fusionsproteine modifizieren oder deren Interaktion vermitteln oder stören.
Die Co-Expression des dritten Proteins kann zur Modifikation oder Aktivierung eines oder beider Fusionsproteine erforderlich sein. Beispielsweise besitzt S. cerevisiae keine endogene Tyrosinkinase. Handelt es sich bei einer Untersuchung um ein Protein, das eine Tyrosin-Phosphorylierung erfordert, muss die Kinase in Form eines Tyrosin-Kinase-Gens geliefert werden.
Das Nicht-Fusionsprotein kann die Wechselwirkung vermitteln, indem es beide Fusionsproteine gleichzeitig bindet, wie im Fall der ligandenabhängigen Rezeptordimerisierung.
Für ein Protein mit einem Wechselwirkungspartner kann seine funktionelle Homologie zu anderen Proteinen bewertet werden, indem das dritte Protein in Nichtfusionsform bereitgestellt wird, das dann mit dem Fusionsprotein um seinen Bindungspartner konkurrieren kann oder nicht. Die Bindung zwischen dem dritten Protein und dem anderen Fusionsprotein wird die Bildung des Reporter-Expressions-Aktivierungskomplexes unterbrechen und somit die Reporter-Expression reduzieren, was zu der charakteristischen Veränderung des Phänotyps führt.
Split-Ubiquitin-Hefe-Zwei-Hybrid
Eine Einschränkung klassischer Hefe-Zwei-Hybrid-Screens besteht darin, dass sie auf lösliche Proteine beschränkt sind. Daher ist es unmöglich, sie zur Untersuchung der Protein-Protein-Wechselwirkungen zwischen unlöslichen integralen Membranproteinen zu verwenden . Das Split-Ubiquitin-System bietet ein Verfahren zur Überwindung dieser Einschränkung. Im Split-Ubiquitin-System werden zwei zu untersuchende integrale Membranproteine an zwei verschiedene Ubiquitin- Einheiten fusioniert : eine C-terminale Ubiquitin-Einheit ("Cub", Reste 35–76) und eine N-terminale Ubiquitin-Einheit ("Nub", Reste 1–34). Diese fusionierten Proteine werden als Köder bzw. Beute bezeichnet. Die Cub-Einheit ist nicht nur an ein integrales Membranprotein fusioniert, sondern auch an einen Transkriptionsfaktor (TF), der von Ubiquitin-spezifischen Proteasen abgespalten werden kann . Bei der Köder-Beute-Interaktion lagern sich Nub- und Cub-Einheiten zusammen und rekonstituieren das gespaltene Ubiquitin. Das rekonstituierte Split-Ubiquitin-Molekül wird von Ubiquitin-spezifischen Proteasen erkannt, die den Transkriptionsfaktor abspalten, wodurch er die Transkription von Reportergenen induzieren kann .
Fluoreszenz-Zwei-Hybrid-Assay
Zolghadr und Mitarbeiter präsentierten ein fluoreszierendes Zwei-Hybrid-System, das zwei Hybridproteine verwendet, die an verschiedene fluoreszierende Proteine fusioniert sind, sowie LacI, den lac-Repressor . Die Struktur der Fusionsproteine sieht so aus: FP2-LacI-bait und FP1-prey, wobei die Köder- und Beuteproteine interagieren und die fluoreszierenden Proteine (FP1 = GFP , FP2 = mCherry ) an der Bindungsstelle des LacI . in enge Nachbarschaft bringen Protein im Genom der Wirtszelle. Das System kann auch verwendet werden, um nach Inhibitoren von Protein-Protein-Wechselwirkungen zu suchen.
Enzymatische Zwei-Hybrid-Systeme: KISS
Während das ursprüngliche Y2H-System einen rekonstituierten Transkriptionsfaktor verwendet, erzeugen andere Systeme enzymatische Aktivitäten zum Nachweis von PPIs. Zum Beispiel ist der KInase Substrate Sensor ("KISS") ein Säugetier-Zwei-Hybrid-Ansatz, der entwickelt wurde, um intrazelluläre PPIs zu kartieren. Dabei wird ein Köderprotein zu einem verschmolzenen kinase -haltigen Anteil TYK2 und Beute auf einem gekoppelten gp130 Cytokinrezeptor Fragment. Wenn Köder und Beute interagieren, phosphoryliert TYK2 STAT3- Andockstellen auf der Beutechimäre , was letztendlich zur Aktivierung eines Reportergens führt .
Ein-, Drei- und Eins-Zwei-Hybrid-Varianten
Ein-Hybrid
Die Ein-Hybrid-Variante dieser Technik wurde entwickelt, um Protein-DNA-Wechselwirkungen zu untersuchen und verwendet ein einziges Fusionsprotein, in dem die AD direkt an die Bindungsdomäne gebunden ist. Die Bindungsdomäne hat in diesem Fall jedoch nicht unbedingt eine feste Sequenz wie bei der Zwei-Hybrid-Protein-Protein-Analyse, sondern kann aus einer Bibliothek bestehen. Diese Bibliothek kann gegen die gewünschte Zielsequenz selektiert werden, die in die Promotorregion des Reportergenkonstrukts eingefügt wird. In einem Positiv-Selektionssystem wird somit eine Bindungsdomäne ausgewählt, die erfolgreich die UAS bindet und die Transkription ermöglicht.
Beachten Sie, dass die Auswahl von DNA-Bindungsdomänen nicht notwendigerweise unter Verwendung eines Ein-Hybrid-Systems durchgeführt wird, sondern auch unter Verwendung eines Zwei-Hybrid-Systems durchgeführt werden kann, in dem die Bindungsdomäne variiert und die Köder- und Beuteproteine konstant gehalten werden.
Drei-Hybrid
RNA-Protein-Interaktionen wurden durch eine Drei-Hybrid-Variante der Zwei-Hybrid-Technik untersucht. In diesem Fall dient ein hybrides RNA-Molekül dazu, die beiden Proteinfusionsdomänen – die nicht miteinander interagieren sollen, sondern das intermediäre RNA-Molekül (über ihre RNA-Bindungsdomänen) – miteinander zu verbinden. Techniken mit Nicht-Fusionsproteinen, die eine ähnliche Funktion erfüllen, wie oben im Abschnitt "Nicht-Fusionsproteine" beschrieben, können auch als Drei-Hybrid-Verfahren bezeichnet werden.
Eins-Zwei-Hybrid
Die gleichzeitige Verwendung der Ein- und Zwei-Hybrid-Methode (d. h. gleichzeitige Protein-Protein- und Protein-DNA-Interaktion) ist als Eins-Zwei-Hybrid-Ansatz bekannt und soll die Stringenz des Screenings erhöhen.
Wirtsorganismus
Obwohl theoretisch jede lebende Zelle als Hintergrund für eine Zwei-Hybrid-Analyse verwendet werden könnte, gibt es praktische Erwägungen, die die Wahl diktieren. Die gewählte Zelllinie sollte relativ billig und leicht zu kultivieren und ausreichend robust sein, um der Anwendung der Untersuchungsmethoden und Reagenzien standzuhalten. Letzteres ist besonders wichtig für die Durchführung von Hochdurchsatzstudien . Daher war die Hefe S. cerevisiae der Hauptwirtsorganismus für Zwei-Hybrid-Studien. Es ist jedoch nicht immer das ideale System, um interagierende Proteine anderer Organismen zu untersuchen. Hefezellen weisen oft nicht die gleichen posttranslationalen Modifikationen auf, haben eine andere Codonverwendung oder es fehlen bestimmte Proteine, die für die korrekte Expression der Proteine wichtig sind. Um diese Probleme zu bewältigen, wurden mehrere neuartige Zwei-Hybrid-Systeme entwickelt. Je nach verwendetem System werden Agarplatten oder ein spezifisches Wachstumsmedium verwendet, um die Zellen zu züchten und eine Selektion auf Interaktion zu ermöglichen. Die am häufigsten verwendete Methode ist die Agar-Plattierung, bei der Zellen auf selektivem Medium plattiert werden, um zu sehen, dass eine Wechselwirkung stattfindet. Zellen, die keine Interaktionsproteine aufweisen, sollten auf diesem selektiven Medium nicht überleben.
S. cerevisiae (Hefe)
Die Hefe S. cerevisiae war der Modellorganismus, der während der Einführung der Zwei-Hybrid-Technik verwendet wurde. Es ist allgemein als Y2H-System bekannt. Es hat mehrere Eigenschaften, die es zu einem robusten Organismus für die Interaktion machen, darunter die Fähigkeit, tertiäre Proteinstrukturen zu bilden, einen neutralen internen pH-Wert, eine verbesserte Fähigkeit zur Bildung von Disulfidbrücken und Glutathion im reduzierten Zustand neben anderen zytosolischen Pufferfaktoren, um ein gastfreundliches internes aufrechtzuerhalten Umgebung. Das Hefemodell kann durch nichtmolekulare Techniken manipuliert werden und seine vollständige Genomsequenz ist bekannt. Hefesysteme sind tolerant gegenüber verschiedenen Kulturbedingungen und aggressiven Chemikalien, die nicht auf Säugetiergewebekulturen angewendet werden könnten.
Eine Reihe von Hefestämmen wurde speziell für Y2H- Screenings erzeugt , zB Y187 und AH109 , die beide von Clontech hergestellt werden . Auch Hefestämme R2HMet und BK100 wurden verwendet.
Candida albicans
C. albicans ist eine Hefe mit einer besonderen Eigenschaft: Sie übersetzt das CUG-Codon in Serin statt in Leucin. Aufgrund dieser unterschiedlichen Codonverwendung ist es schwierig, das Modellsystem S. cerevisiae als Y2H zu verwenden, um Protein-Protein-Interaktionen mit C. albicans- Genenzu überprüfen. Um einenativereUmgebung bereitzustellen, wurde ein C. albicans- Zwei-Hybrid (C2H)-System entwickelt. Mit diesem System können Protein-Protein-Wechselwirkungen in C. albicans selbst untersucht werden. Neu hinzugekommen ist die Schaffung eines Hochdurchsatzsystems.
E coli
Bakterielle Zwei-Hybrid-Methoden (B2H oder BTH) werden normalerweise in E. coli durchgeführt und haben einige Vorteile gegenüber hefebasierten Systemen. Zum Beispiel verleihen die höhere Transformationseffizienz und die schnellere Wachstumsrate E. coli die Verwendung größerer Bibliotheken (über 10 8 ). Das Fehlen von Anforderungen für die Aufnahme eines nuklearen Lokalisierungssignals in die Proteinsequenz und die Fähigkeit, Proteine zu untersuchen, die für Hefe toxisch wären, können ebenfalls wichtige Faktoren sein, die bei der Auswahl eines experimentellen Hintergrundorganismus zu berücksichtigen sind.
Die Methylierungsaktivität bestimmter E. coli- DNA-Methyltransferase- Proteine kann die Auswahl einiger DNA-bindender Proteine beeinträchtigen. Wenn dies erwartet wird, kann die Verwendung eines E. coli- Stammes, der für eine bestimmte Methyltransferase defekt ist, eine naheliegende Lösung sein. Der B2H ist möglicherweise nicht ideal, wenn eukaryotische Protein-Protein-Interaktionen (z. B. menschliche Proteine) untersucht werden, da Proteine möglicherweise nicht wie in eukaryotischen Zellen falten oder andere Verarbeitungsschritte fehlen.
Säugetierzellen
In den letzten Jahren wurde ein Säugetier-Zwei-Hybrid-System (M2H) entwickelt, um Protein-Protein-Interaktionen von Säugetieren in einer zellulären Umgebung zu untersuchen, die der nativen Proteinumgebung sehr nahe kommt. Transient transfizierte Säugerzellen werden in diesem System verwendet, um Protein-Protein-Wechselwirkungen zu finden. Die Verwendung einer Säugetierzelllinie zur Untersuchung von Protein-Protein-Interaktionen von Säugetieren bietet den Vorteil, in einem nativeren Kontext zu arbeiten. Die posttranslationalen Modifikationen Phosphorylierung, Acylierung und Glykosylierung sind ähnlich. Die intrazelluläre Lokalisierung der Proteine ist auch im Vergleich zur Verwendung eines Hefe-Zwei-Hybrid-Systems korrekter. Mit dem Säugetier-Zwei-Hybrid-System ist es auch möglich, Signaleingänge zu untersuchen. Ein weiterer großer Vorteil ist, dass die Ergebnisse innerhalb von 48 Stunden nach der Transfektion erhalten werden können.
Arabidopsis thaliana
2005 wurde ein Zwei-Hybrid-System in Anlagen entwickelt. Mithilfe von Protoplasten von A. thaliana können Protein-Protein-Wechselwirkungen in Pflanzen untersucht werden. Auf diese Weise können die Interaktionen in ihrem ursprünglichen Kontext untersucht werden. In diesem System stehen GAL4 AD und BD unter der Kontrolle des starken 35S-Promotors. Die Interaktion wird mit einem GUS-Reporter gemessen. Um ein Hochdurchsatz-Screening zu ermöglichen, wurden die Vektoren Gateway-kompatibel gemacht. Das System ist als Protoplasten-Zwei-Hybrid-(P2H)-System bekannt.
Aplysia californica
Der Seehase A californica ist ein Modellorganismus in der Neurobiologie, um unter anderem die molekularen Mechanismen des Langzeitgedächtnisses zu untersuchen. Um Interaktionen, die in der Neurologie wichtig sind, in einer nativeren Umgebung zu untersuchen, wurde ein Zwei-Hybrid-System in A californica- Neuronen entwickelt. In diesem System werden ein GAL4 AD und BD verwendet.
Bombyx mori
Ein Insekten-Zwei-Hybrid (I2H)-System wurde in einer Seidenraupenzelllinie aus der Larve oder Raupe der domestizierten Seidenmotte Bombyx mori (BmN4-Zellen) entwickelt. Dieses System verwendet die GAL4 BD und die Aktivierungsdomäne von Maus-NF-κB P65. Beide stehen unter der Kontrolle des OpIE2-Promotors.
Anwendungen
Bestimmung der für die Interaktion entscheidenden Sequenzen
Durch Veränderung spezifischer Aminosäuren durch Mutation der entsprechenden DNA-Basenpaare in den verwendeten Plasmiden kann die Bedeutung dieser Aminosäurereste für die Aufrechterhaltung der Wechselwirkung bestimmt werden.
Nach der Verwendung einer auf Bakterienzellen basierenden Methode zur Auswahl von DNA-bindenden Proteinen muss die Spezifität dieser Domänen überprüft werden, da das Genom der Bakterienzelle als Senke für Domänen mit einer Affinität zu anderen fungieren kann Sequenzen (oder tatsächlich eine allgemeine Affinität für DNA).
Entdeckung von Drogen und Giften
Protein-Protein-Signalwechselwirkungen stellen aufgrund ihrer Spezifität und Verbreitung geeignete therapeutische Angriffspunkte dar. Der Ansatz der zufälligen Wirkstoffforschung verwendet Verbindungsbanken, die zufällige chemische Strukturen umfassen, und erfordert eine Hochdurchsatzmethode, um diese Strukturen in ihrem beabsichtigten Ziel zu testen.
Die für die Untersuchung ausgewählte Zelle kann speziell so konstruiert werden, dass sie den molekularen Aspekt widerspiegelt, den der Forscher untersuchen möchte, und dann verwendet werden, um neue menschliche oder tierische Therapeutika oder Schädlingsbekämpfungsmittel zu identifizieren.
Bestimmung der Proteinfunktion
Durch die Bestimmung der Interaktionspartner unbekannter Proteine kann auf die möglichen Funktionen dieser neuen Proteine geschlossen werden. Dies kann unter Verwendung eines einzelnen bekannten Proteins gegen eine Bibliothek unbekannter Proteine oder umgekehrt durch Auswahl aus einer Bibliothek bekannter Proteine unter Verwendung eines einzelnen Proteins unbekannter Funktion erfolgen.
Auswahl von Zinkfingerproteinen
Um Zinkfingerproteine (ZFPs) für das Protein-Engineering zu selektieren , wurden mit Erfolg Methoden verwendet, die von der Zwei-Hybrid-Screening-Technik adaptiert wurden. Ein ZFP ist selbst ein DNA-bindendes Protein, das bei der Konstruktion von benutzerdefinierten DNA-bindenden Domänen verwendet wird, die an eine gewünschte DNA-Sequenz binden.
Durch Verwendung eines Selektionsgens mit der gewünschten Zielsequenz, das in der UAS enthalten ist, und Randomisierung der relevanten Aminosäuresequenzen, um eine ZFP-Bibliothek herzustellen, können Zellen selektiert werden, die eine DNA-ZFP-Interaktion mit den erforderlichen Eigenschaften beherbergen. Jedes ZFP erkennt normalerweise nur 3–4 Basenpaare. Um die Erkennung von Stellen außerhalb der UAS zu verhindern, wird das randomisierte ZFP in ein „Gerüst“ eingearbeitet, das aus weiteren zwei ZFPs mit konstanter Sequenz besteht. Der UAS ist somit so ausgelegt, dass er zusätzlich zu der Sequenz, für die ein ZFP ausgewählt wird, die Zielsequenz des konstanten Gerüsts enthält.
Mit diesem System können auch eine Reihe anderer DNA-bindender Domänen untersucht werden.
Stärken
- Zwei-Hybrid-Bildschirme sind Low-Tech; sie können in jedem Labor ohne hoch entwickelte Geräte durchgeführt werden.
- Zwei-Hybrid-Screens können einen wichtigen ersten Hinweis zur Identifizierung von Interaktionspartnern geben.
- Der Assay ist skalierbar, was es ermöglicht, auf Wechselwirkungen zwischen vielen Proteinen zu screenen. Darüber hinaus kann es automatisiert werden, und durch den Einsatz von Robotern können viele Proteine in relativ kurzer Zeit gegen Tausende von potenziell interagierenden Proteinen gescreent werden. Zwei Arten von Großbildschirmen werden verwendet: der Bibliotheksansatz und der Matrixansatz.
- Hefe-Zwei-Hybrid-Daten können von ähnlicher Qualität sein wie Daten, die durch den alternativen Ansatz der Koaffinitätsreinigung gefolgt von Massenspektrometrie (AP/MS) erzeugt werden.
Schwächen
- Der Hauptkritikpunkt beim Hefe-Zwei-Hybrid-Screening von Protein-Protein-Wechselwirkungen ist die Möglichkeit einer hohen Anzahl falsch positiver (und falsch negativer) Identifizierungen. Die genaue Rate falsch positiver Ergebnisse ist nicht bekannt, aber frühere Schätzungen lagen bei bis zu 70 %. Dies erklärt zum Teil auch die oft gefundene sehr geringe Überlappung der Ergebnisse beim Einsatz eines (Hochdurchsatz-)Zwei-Hybrid-Screenings, insbesondere bei Verwendung unterschiedlicher Versuchssysteme.
Der Grund für diese hohe Fehlerquote liegt in den Eigenschaften des Bildschirms:
- Bestimmte Testvarianten überexprimieren die Fusionsproteine, was zu unnatürlichen Proteinkonzentrationen führen kann, die zu unspezifischen (falsch) positiven Ergebnissen führen.
- Die Hybridproteine sind Fusionsproteine; das heißt, die fusionierten Teile können bestimmte Interaktionen hemmen, insbesondere wenn eine Interaktion am N-Terminus eines Testproteins stattfindet (wo die DNA-Bindungs- oder Aktivierungsdomäne typischerweise angebracht ist).
- In Hefe, dem typischen Wirtsorganismus für Y2H, darf keine Interaktion stattfinden. Wenn beispielsweise ein bakterielles Protein in Hefe getestet wird, fehlt ihm möglicherweise ein Chaperon für die richtige Faltung, das nur in seinem bakteriellen Wirt vorhanden ist. Darüber hinaus wird ein Säugerprotein in Hefe manchmal nicht korrekt modifiziert (zB fehlende Phosphorylierung ), was ebenfalls zu falschen Ergebnissen führen kann.
- Das Y2H findet im Kern statt. Wenn Testproteine nicht im Zellkern lokalisiert sind (weil sie andere Lokalisierungssignale haben), kann festgestellt werden, dass zwei interagierende Proteine nicht interagieren.
- Einige Proteine können spezifisch interagieren, wenn sie in der Hefe co-exprimiert werden, obwohl sie in Wirklichkeit nie gleichzeitig in derselben Zelle vorhanden sind. In den meisten Fällen kann jedoch nicht ausgeschlossen werden, dass solche Proteine in bestimmten Zellen oder unter Umständen tatsächlich exprimiert werden.
Jeder dieser Punkte allein kann zu falschen Ergebnissen führen. Aufgrund der kombinierten Effekte aller Fehlerquellen ist Hefe-Zwei-Hybrid mit Vorsicht zu interpretieren. Die Wahrscheinlichkeit der Erzeugung von falschen Positiven bedeuten , daß alle Wechselwirkungen sollten von einem hohen Vertrauenstest bestätigt werden, beispielsweise Co-Immunpräzipitation der endogenen Proteine, die für große Protein-Protein - Interaktionsdaten schwierig ist. Alternativ können Y2H-Daten mit mehreren Y2H-Varianten oder bioinformatischen Techniken verifiziert werden. Letztere testen, ob wechselwirkende Proteine gleichzeitig exprimiert werden, einige Gemeinsamkeiten aufweisen (wie genontologische Annotationen oder bestimmte Netzwerktopologien ), homologe Wechselwirkungen in anderen Spezies aufweisen.
Siehe auch
- Phagen-Display , eine alternative Methode zum Nachweis von Protein-Protein- und Protein-DNA-Wechselwirkungen
- Protein-Array , eine chipbasierte Methode zum Nachweis von Protein-Protein-Wechselwirkungen
- Synthetische genetische Array- Analyse, eine hefebasierte Methode zur Untersuchung von Geninteraktionen
Verweise
Externe Links
|
Bibliotheksressourcen zum Two-Hybrid-Screening |
- Detail zum Schwestertechnik-Zwei-Hybrid-System
- Überblick von Science Creative Quarterly über das Hefe-Zwei-Hybrid-System
- Gateway-kompatible Hefe-One-Hybrid-Bildschirme
- Videoanimation des Yeast Two-Hybrid Systems
- Two-Hybrid+System+Techniques in der US National Library of Medicine Medical Subject Headings (MeSH)
- Hefe-Zwei-Hybrid
- BioGrid-Datenbank mit Protein-Protein-Interaktionen

