Biomarker des Alterns - Biomarkers of aging
Biomarker des Alterns sind Biomarker , die die funktionelle Leistungsfähigkeit in einem späteren Alter besser vorhersagen können als im chronologischen Alter. Anders ausgedrückt würden Biomarker des Alterns das wahre "biologische Alter" ergeben, das sich vom chronologischen Alter unterscheiden kann.
Validierte Biomarker des Alterns würden es ermöglichen, Interventionen zur Verlängerung der Lebensdauer zu testen , da Veränderungen der Biomarker während der gesamten Lebensdauer des Organismus beobachtbar wären. Obwohl die maximale Lebensdauer ein Mittel zur Validierung von Biomarkern des Alterns wäre, wäre es für langlebige Arten wie den Menschen kein praktikables Mittel, da Längsschnittstudien viel zu viel Zeit in Anspruch nehmen würden. Im Idealfall sollten Biomarker des Alterns den biologischen Alterungsprozess und keine Prädisposition für Krankheiten untersuchen, sollten ein minimales Ausmaß an Trauma beim Testen im Organismus verursachen und sollten während eines kurzen Intervalls im Vergleich zur Lebensdauer des Organismus reproduzierbar messbar sein. Eine Ansammlung von Biomarker-Daten für einen Organismus könnte als sein "Ageotyp" bezeichnet werden.
Obwohl das Ergrauen der Haare mit dem Alter zunimmt, kann das Ergrauen der Haare nicht als Biomarker für das Altern bezeichnet werden. Ebenso sind Hautfalten und andere häufige Veränderungen, die mit dem Altern beobachtet werden, keine besseren Indikatoren für die zukünftige Funktionalität als das chronologische Alter. Biogerontologen haben sich weiterhin bemüht, Biomarker des Alterns zu finden und zu validieren, aber der Erfolg war bisher begrenzt. Die Konzentrationen von CD4- und CD8- Gedächtnis-T-Zellen und naiven T-Zellen wurden verwendet, um gute Vorhersagen über die erwartete Lebensdauer von Mäusen mittleren Alters zu treffen.
Fortschritte in der Big-Data- Analyse ermöglichten die Entwicklung neuer Arten von „Alterungsuhren“. Die epigenetische Uhr ist ein vielversprechender Biomarker für das Altern und kann das chronologische Alter des Menschen genau vorhersagen. Die grundlegende Blutbiochemie und Zellzahlen können auch verwendet werden, um das chronologische Alter genau vorherzusagen. Weitere Studien der hämatologischen Uhr an den großen Datensätzen südkoreanischer, kanadischer und osteuropäischer Bevölkerungen zeigten, dass Biomarker des Alterns bevölkerungsspezifisch und prädiktiv für die Sterblichkeit sein können. Es ist auch möglich, das chronologische Alter des Menschen mit Hilfe der transkriptomischen Uhr vorherzusagen .
Die jüngste Einführung von stromsparenden und kompakten Sensoren, die auf mikroelektromechanischen Systemen ( MEMS ) basieren, hat zu einer neuen Generation tragbarer und erschwinglicher Geräte geführt, die beispiellose Möglichkeiten zum Sammeln und Speichern persönlicher digitalisierter Aktivitätsaufzeichnungen bieten. Folglich könnten moderne Techniken des tiefen maschinellen Lernens verwendet werden, um einen konzeptionellen digitalen Biomarker für das Alter in Form eines All-Ursachen-Mortalitäts-Prädiktors aus einer ausreichend großen Sammlung von einwöchigen menschlichen Bewegungsströmen zu erstellen, die durch die reichhaltigen klinischen Daten (einschließlich des Sterberegisters, wie es z. B. von der NHANES- Studie bereitgestellt wird ).
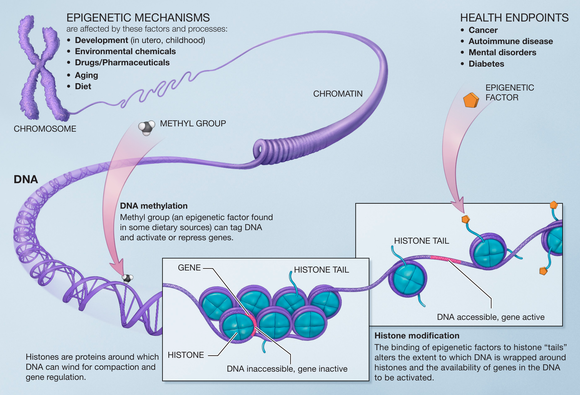
Epigenetische Markierungen
Verlust von Histonen
Ein neues epigenetisches Merkmal, das in Studien alternder Zellen gefunden wurde, ist der Verlust von Histone . Die meisten Beweise zeigen, dass der Verlust von Histone mit der Zellteilung verbunden ist. In alternden und sich teilenden Hefen zeigte MNase-seq (Micrococcal Nuclease Sequencing) einen Verlust an Nukleosomen von ~50%. Die richtige Histondosierung ist bei Hefe wichtig, wie die verlängerte Lebensdauer von Stämmen zeigt, die Histone überexprimieren. Eine Folge des Histonverlustes in Hefe ist die Amplifikation der Transkription . In jüngeren Zellen, die Gene , die meisten sind mit dem Alter haben Strukturen spezifische Chromatin induziert, wie beispielsweise fuzzy Kernpositionierung, das Fehlen eines nuclesome verarmten Bereich (NDR) am Promotor , schwach Chromatin Phasing, eine höhere Frequenz der TATA - Elemente , und eine höhere Belegung repressive Chromatinfaktoren. In älteren Zellen ist jedoch der Nukleosomenverlust der gleichen Gene am Promotor häufiger, was zu einer höheren Transkription dieser Gene führt.
Dieses Phänomen wird nicht nur bei Hefe beobachtet, sondern wurde auch bei alternden Würmern, während der Alterung von menschlichen diploiden primären Fibroblasten und bei seneszenten menschlichen Zellen beobachtet. In humanen primären Fibroblasten wurde die reduzierte Synthese neuer Histone als Folge verkürzter Telomere gesehen , die die Reaktion auf DNA-Schäden aktivieren. Der Verlust von Kernhistonen kann ein allgemeines epigenetisches Zeichen des Alterns bei vielen Organismen sein.
Histon-Varianten
Neben den Kernhistonen H2A, H2B, H3 und H4 gibt es weitere Versionen der Histonproteine, die sich in ihrer Sequenz deutlich unterscheiden können und für die Regulierung der Chromatindynamik wichtig sind. Histon H3.3 ist eine Variante von Histon H3, die unabhängig von der Replikation in das Genom eingebaut wird. Es ist die Hauptform von Histon H3, die im Chromatin seneszenter menschlicher Zellen vorkommt, und es scheint, dass überschüssiges H3.3 die Seneszenz fördern kann .
Es gibt mehrere Varianten von Histon 2, die am stärksten mit dem Altern in Verbindung gebrachte ist MakroH2A. Die Funktion von MacroH2A wird allgemein als transkriptionelles Silencing angenommen; vor kurzem wurde vorgeschlagen, dass MacroH2A bei der Unterdrückung der Transkription an Seneszenz-assoziierten Heterochromatin-Foci (SAHF) wichtig ist. Chromatin, das MacroH2A enthält, ist für ATP-abhängige Remodeling-Proteine und die Bindung von Transkriptionsfaktoren unempfindlich .
Histon-Modifikationen
Eine erhöhte Acetylierung von Histonen trägt dazu bei, dass das Chromatin mit zunehmendem Alter eines Organismus einen euchromatischeren Zustand einnimmt, ähnlich der erhöhten Transkription , die aufgrund des Verlustes von Histonen beobachtet wird. Es gibt auch eine Verringerung der Konzentrationen von H3K56ac während des Alterns und eine Erhöhung des Niveaus von H4K16ac . Erhöhte H4K16ac-Werte in alten Hefezellen sind mit der Abnahme des HDAC- Sir2- Spiegels verbunden , der bei Überexpression die Lebensdauer verlängern kann.
Die Methylierung von Histonen wurde in vielen Organismen mit der Regulierung der Lebensspanne in Verbindung gebracht, insbesondere H3K4me3, eine aktivierende Markierung, und H4K27me3, eine unterdrückende Markierung. Bei C. elegans senkt der Verlust eines der drei Trithorax-Proteine, die die Trimethylierung von H3K4 katalysieren, wie WDR-5 und die Methyltransferasen SET-2 und ASH-2, den H3K4me3-Spiegel und verlängert die Lebensdauer. Der Verlust des Enzyms, das H3K4me3 demethyliert, RB-2, erhöht den H3K4me3-Spiegel in C. elegans und verkürzt deren Lebensdauer. In dem Rhesusaffe Gehirn präfrontalen Kortex , H3K4me2 steigt an Promotor und Enhancer während der postnatalen Entwicklung und Alterung . Diese Zunahmen spiegeln zunehmend aktivere und transkriptionell zugängliche (oder offene) Chromatinstrukturen wider , die oft mit Stressreaktionen wie der DNA-Schadensreaktion verbunden sind . Diese Veränderungen können eine epigenetische Erinnerung an Belastungen und Schäden bilden, die der Organismus während seiner Entwicklung und seines Alterns erfährt.
UTX-1, eine H3K27me3- Demethylase, spielt eine entscheidende Rolle bei der Alterung von C.elegans : Eine erhöhte utx-1- Expression korreliert mit einer Abnahme von H3K27me3 und einer Abnahme der Lebensdauer. Utx-1- Knockdowns zeigten eine Verlängerung der Lebensdauer Veränderungen des H3K27me3-Spiegels wirken sich auch auf alternde Zellen bei Drosophila und Menschen aus.
DNA-Methylierung
Die Methylierung von DNA ist eine häufige Modifikation in Säugerzellen . Die Cytosinbase ist methyliert und wird zu 5-Methylcytosin , am häufigsten im CpG- Kontext. Eine Hypermethylierung von CpG-Inseln ist mit einer transkriptionalen Repression verbunden und eine Hypomethylierung dieser Stellen ist mit einer transkriptionalen Aktivierung verbunden. Viele Studien haben gezeigt, dass es bei vielen Arten wie Ratten, Mäusen, Kühen, Hamstern und Menschen während des Alterns zu einem Verlust der DNA-Methylierung kommt. Es wurde auch gezeigt, dass DNMT1 und DNMT3a mit zunehmendem Alter abnehmen und DNMT3b zunimmt.
Die Hypomethylierung der DNA kann die genomische Stabilität verringern, die Reaktivierung von transponierbaren Elementen induzieren und den Verlust der Prägung verursachen , was alle zur Krebsprogression und Pathogenese beitragen kann .
Immunbiomarker
Jüngste Daten legen nahe, dass eine erhöhte Häufigkeit von seneszenten CD8+ T-Zellen im peripheren Blut mit der Entwicklung einer Hyperglykämie aus einem prädiabetischen Zustand verbunden ist, was darauf hindeutet, dass Seneszenz eine Rolle bei der metabolischen Alterung spielt. Seneszente Cd8+-T-Zellen könnten als Biomarker verwendet werden, um den Übergang von Prädiabetes zu manifester Hyperglykämie zu signalisieren.
Kürzlich haben Hashimoto und Mitarbeiter Tausende von zirkulierenden Immunzellen von Supercentenarians mit Einzelzellauflösung profiliert. Sie identifizierten eine sehr einzigartige Zunahme zytotoxischer CD4-T-Zellen bei diesen über Hundertjährigen. Im Allgemeinen haben CD4-T-Zellen unter physiologischen Bedingungen Helfer-, aber nicht zytotoxische Funktionen, jedoch zeigten diese Supercentenarians, die einem Einzelzell-Profiling ihrer T-Zell-Rezeptoren unterzogen wurden, Ansammlungen von zytotoxischen CD4-T-Zellen durch klonale Expansion. Die Umwandlung von CD4-Helferzellen in eine zytotoxische Sorte könnte eine Anpassung an das späte Alterungsstadium sein, die bei der Bekämpfung von Infektionen hilft und möglicherweise die Tumorüberwachung verbessert.
Anwendungen von alternden Biomarkern
Die wichtigsten Mechanismen, die als potenzielle Biomarker des Alterns identifiziert wurden, sind DNA-Methylierung, Verlust von Histonen und Histonmodifikation. Die Verwendung von Biomarkern des Alterns ist allgegenwärtig und die Identifizierung eines physikalischen Parameters des biologischen Alterns würde es dem Menschen ermöglichen, unser wahres Alter, unsere Sterblichkeit und Morbidität zu bestimmen. Die Veränderung des physikalischen Biomarkers sollte proportional zur Veränderung des Alters der Spezies sein. So könnten Menschen nach der Etablierung eines Biomarkers für das Altern in die Forschung zur Verlängerung der Lebensspanne eintauchen und Zeitpläne für das Auftreten potenzieller genetischer Krankheiten finden.
Eine der Anwendungen dieses Ergebnisses würde die Identifizierung des biologischen Alters einer Person ermöglichen. DNA-Methylierung verwendet die Struktur der DNA in verschiedenen Lebensphasen, um ein Alter zu bestimmen. DNA-Methylierung ist die Methylierung des Cysteins in der CG- oder Cpg-Region. Die Hypermethylierung dieser Region ist mit einer verminderten Transkriptionsaktivität und das Gegenteil für eine Hypomethylierung verbunden. Mit anderen Worten, je "fester" die DNA-Region gehalten wird, desto stabiler und "jünger" ist die Spezies. Betrachtet man die Eigenschaften der DNA-Methylierung in Geweben, wurde festgestellt, dass sie für embryonale Gewebe fast Null ist, sie kann verwendet werden, um die Beschleunigung des Alters zu bestimmen, und die Ergebnisse können in Schimpansengewebe reproduziert werden.