U2-spleißosomale RNA - U2 spliceosomal RNA
| U2-spleißosomale RNA | |
|---|---|
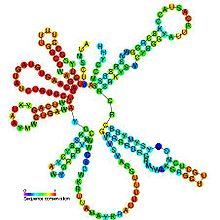 Vorhergesagte Sekundärstruktur- und Sequenzerhaltung von U2
| |
| Bezeichner | |
| Symbol | U2 |
| Rfam | RF00004 |
| Andere Daten | |
| RNA- Typ | Gen ; snRNA ; Spleißen |
| Domain(s) | Eukaryoten |
| GEHEN | GO:0000370 GO:0045131 GO:0005686 |
| SO | SO:0000392 |
| PDB- Strukturen | PDBe |
U2-spleißosomale snRNAs sind eine Spezies kleiner nukleärer RNA- Moleküle ( snRNA ), die in der wichtigsten spleißosomalen (Sm) Maschine praktisch aller eukaryotischen Organismen vorkommen. In vivo assemblieren U2-snRNA zusammen mit ihren assoziierten Polypeptiden, um das kleine nukleäre U2 -Ribonukleoprotein ( snRNP ) zu produzieren, eine wesentliche Komponente des Hauptspleißosomkomplexes. Der Hauptspleißosom-Spleißweg wird gelegentlich als U2-abhängig bezeichnet, basierend auf einer Klasse von Sm- Introns – die in primären mRNA-Transkripten zu finden sind –, die während der frühen Stadien der spleißosomalen Assemblierung ausschließlich vom U2-snRNP erkannt werden. Neben der U2-abhängigen Intron-Erkennung wird U2-snRNA theoretisch auch eine katalytische Rolle in der Chemie des Prä-RNA-Spleißens zugeschrieben. Ähnlich wie ribosomale RNAs ( rRNAs ) müssen Sm-snRNAs sowohl RNA:RNA- als auch RNA:Protein-Kontakte vermitteln und haben daher spezialisierte, hochkonservierte, primäre und sekundäre Strukturelemente entwickelt, um diese Arten von Interaktionen zu ermöglichen.
Kurz nach der Entdeckung von Sharp und Roberts , dass mRNA- Primärtranskripte lange, nicht-kodierende Zwischensequenzen ( Introns ) enthalten, begann Joan Steitz mit der Charakterisierung des biochemischen Mechanismus der Intron-Exzision. Die merkwürdige Beobachtung, dass eine Sequenz, die in der 5´-Region der U1-snRNA gefunden wurde, eine umfangreiche Basenpaarungskomplementarität mit konservierten Sequenzen über 5´-Spleißstellen in hnRNA- Transkripten aufwies, führte zu Spekulationen, dass bestimmte snRNAs an der Erkennung von Spleißstellengrenzen durch RNA:RNA-Kontakte beteiligt sein könnten . Erst kürzlich haben atomare Kristallstrukturen nachweislich gezeigt, dass die ursprüngliche Vermutung tatsächlich richtig war, auch wenn die Komplexität dieser Wechselwirkungen damals noch nicht vollständig erkannt wurde.
U2 snRNA-Erkennungselemente
In Saccharomyces cerevisiae ist die U2-snRNA mit 18 Polypeptiden assoziiert , von denen sieben strukturelle Proteine sind, die allen snRNPs der Sm-Klasse gemeinsam sind. Diese unspezifischen Strukturproteine assoziieren mit Sm-snRNAs durch eine hochkonservierte Erkennungssequenz (AU n G, n = 4-6), die sich innerhalb der RNA befindet, genannt Sm-Bindungsstellen. Zwei weitere Proteine, A´ und B´´, sind U2-spezifisch und benötigen für die snRNP-Assemblierung einzigartige Strukturelemente der U2-snRNA – insbesondere zwei 3´-Stammschleifen. Die Proteinkomplexe aus drei Untereinheiten SF3a und sechs Untereinheiten SF3b assoziieren auch mit der U2-snRNA.
U2 snRNA ist an der Intronerkennung durch eine 7-12 Nukleotidsequenz zwischen 18-40 Nukleotiden stromaufwärts der 3´-Spleißstelle, bekannt als Branch Point Sequence (BPS), beteiligt. In Hefe hat die Konsensus-BPS eine Länge von 7 Nukleotidresten und die komplementäre Erkennungssequenz innerhalb der U2-snRNA ist 6 Nukleotide lang. Die Duplexbildung zwischen diesen beiden Sequenzen führt zur Ausbuchtung eines konservierten Adenosinrestes an Position 5 des BPS. Der gewölbte Adenosinrest nimmt eine C3´-endo-Konformation an, die mit Hilfe der Spleißfaktoren Cwc25, Yju2 und Isy1 ein 2´-OH für einen Inline-Angriff eines Phosphoratoms an der 5´-Spleißstelle ausrichtet . Der nukleophile Angriff initiiert die erste von zwei aufeinanderfolgenden Umesterungsreaktionen , die das Intron herausspleißen – durch ein ungewöhnliches 2´-5´-3´-verknüpftes Lariat-Intermediat – wobei die zweite Umesterung die Ligation der beiden flankierenden Exons beinhaltet.
Primär- und Sekundärstruktur
Obwohl die Sequenzlänge von U2-snRNAs bei allen eukaryontischen Organismen um bis zu einer Größenordnung variieren kann , enthalten alle U2-snRNAs viele phylogenetisch konstante Regionen, insbesondere innerhalb der ersten 80 Nukleotide stromabwärts des 5´-Endes, wo 85% der Positionen konserviert sind. Darüber hinaus sind auch mehrere sekundäre Strukturelemente konserviert, einschließlich der Stammschleifen I, II, III, IV und einige der einzelsträngigen Regionen, die diese Domänen verbinden. Stammschleife II in Hefe-U2-snRNA enthält ein ungewöhnliches geschertes GA-Basenpaar, das zu einem charakteristischen U-Turn-Schleifenmotiv führt, das eine geometrische Konformation ähnlich der von tRNA- Anti-Codon-Schleifen aufweist. Alle U2-snRNAs besitzen eine terminale Stammschleife (IV) mit einer Helix von 10-16 Basenpaaren und eine konservierte 11-Nukleotid-Schleife mit der Konsensussequenz 5´-UYGCANUURYN-3´.
U2-snRNAs sind die am umfangreichsten modifizierten aller kleinen nukleären RNAs. Während die genauen Orte dieser posttranskriptionalen Modifikationen von Organismus zu Organismus variieren können, deuten neue Erkenntnisse darauf hin, dass eine starke Korrelation zwischen der U2-snRNA-Modifikation und der biologischen Funktion besteht. Modifikationen umfassen die Umwandlung einiger Uridinreste in Pseudouridin , 2´-O-Methylierung, Nukleobasenmethylierung und Umwandlung von 5´-monomethylierter Guanosinkappe in eine 2,2,7-trimethylierte Guanosinkappe. Viele dieser Modifikationen befinden sich in einer 27-Nukleotid-Region am 5´-Ende des Moleküls.
Konformationsdynamik
Das Spleißosom ist eine dynamische molekulare Maschine, die während des Zusammenbaus und Spleißens mehrere Konformationsänderungen durchmacht. Obwohl viele der biochemischen Details der spleißosomalen Umlagerung unklar bleiben, haben neuere Studien die Bildung eines kritischen Faltungskomplexes zwischen U2- und U6-snRNAs sichtbar gemacht, der unmittelbar den ersten Schritt der Spleißreaktion vorantreibt. Dieses Faltungsereignis erleichtert die Bildung einer Vier-Helix-Verbindung, von der angenommen wird, dass sie ein Gerüst für die kritischen Komponenten des aktiven Zentrums bietet, einschließlich der Ausrichtung der 5´-Spleißstelle mit dem Verzweigungspunkt Adenosin für den Inline-Angriff durch das 2´-OH und die Koordination von zwei Mg 2+ -Ionen zur Stabilisierung der negativen Ladungsbildung in den Verfahrensschritten.
Evolutionäre Ursprünge
Ein bemerkenswertes Merkmal der U2-U6-Faltung ist ihre strukturelle Ähnlichkeit mit der der Domäne V in selbstspleißenden Gruppe-II-Introns . Die in U6-snRNA gefundene AGC-Triade ist in Gruppe-II-Introns konserviert und begünstigt ebenfalls die gleichen tertiären Stapelwechselwirkungen. Die Bildung eines GU-Wobble-Paares zu Beginn des U2-U6-Faltungsereignisses wird auch bei der Bildung des katalytischen Kerns von Gruppe-II-Introns beobachtet. Schließlich ist es wahrscheinlich, dass das Spleißosom aufgrund der strukturellen Konservierung der Metallbindungsstellen innerhalb der U2-U6-Faltung denselben Zwei-Metall-Ionen-Mechanismus wie Introns der Gruppe II verwendet. Das Ausmaß der Erhaltung der Sekundär- und Tertiärstruktur zwischen Gruppe-II-Introns und der U2-U6-Faltung im aktiven Zentrum des Spleißosoms weist stark darauf hin, dass beide Gruppe-II-Introns und das Spleißosom einen gemeinsamen evolutionären Ursprung haben.
Siehe auch
Verweise
Weiterlesen
- Newby MI, Greenbaum NL (Juni 2001). "Eine konservierte Pseudouridin-Modifikation in eukaryotischer U2-snRNA induziert eine Veränderung der Branch-Site-Architektur" . RNA . 7 (6): 833–45. doi : 10.1017/S1355838201002308 . PMC 1370140 . PMID 11424937 .
- Berglund JA, Rosbash M, Schultz SC (Mai 2001). „Kristallstruktur eines Modells Branchpoint-U2-snRNA-Duplex, das gewölbte Adenosine enthält“ . RNA . 7 (5): 682-91. doi : 10.1017/S1355838201002187 . PMC 1370120 . PMID 11350032 .
