Nukleoid - Nucleoid
Die Nucleoid (Bedeutung Kern -ähnlichen ) ist eine unregelmäßig geformte Region innerhalb der prokaryotischen Zelle , die alle oder die meisten der enthält genetisches Material . Das Chromosom eines Prokaryoten ist kreisförmig und seine Länge ist im Vergleich zu den Zellabmessungen sehr groß, sodass es kompaktiert werden muss, um hineinzupassen. Im Gegensatz zum Kern einer eukaryontischen Zelle ist er nicht von einer Kernmembran umgeben . Stattdessen werden die Nucleoid Formen durch Kondensation und funktionelle Anordnung mit Hilfe von chromosomalen architektonischen Proteinen und RNA - Moleküle sowie DNA supercoiling . Die Länge eines Genoms variiert stark (im Allgemeinen mindestens einige Millionen Basenpaare) und eine Zelle kann mehrere Kopien davon enthalten.
Eine hochaufgelöste Struktur eines bakteriellen Nukleoids ist noch nicht bekannt, jedoch wurden Schlüsselmerkmale an Escherichia coli als Modellorganismus erforscht . In E. coli ist die chromosomale DNA im Durchschnitt negativ supercoiled und in plektonemische Schleifen gefaltet , die auf verschiedene physikalische Regionen beschränkt sind und selten ineinander diffundieren. Diese Schleifen organisieren sich räumlich in Megabasen-große Regionen, die Makrodomänen genannt werden, in denen DNA-Stellen häufig interagieren, zwischen denen jedoch selten Wechselwirkungen auftreten. Die kondensierte und räumlich organisierte DNA bildet ein spiralförmiges Ellipsoid, das radial in der Zelle eingeschlossen ist. Die 3D-Struktur der DNA im Nuceoid scheint abhängig von den Bedingungen zu variieren und ist mit der Genexpression verbunden, so dass die Nukleoidarchitektur und die Gentranskription eng voneinander abhängig sind und sich gegenseitig beeinflussen.

Hintergrund
Bei vielen Bakterien ist das Chromosom ein einzelnes kovalent geschlossenes (zirkuläres) doppelsträngiges DNA-Molekül, das die genetische Information in haploider Form kodiert . Die Größe der DNA variiert zwischen 500.000 und mehreren Millionen Basenpaaren (bp), die je nach Organismus 500 bis mehrere tausend Gene kodieren. Die chromosomale DNA liegt in Zellen in einer sehr kompakten, organisierten Form vor, die als Nukleoid (bedeutet kernartig ) bezeichnet wird und nicht wie in eukaryotischen Zellen von einer Kernmembran umhüllt ist. Das isolierte Nukleoid enthält 80 Gew.-% DNA, 10 Gew.-% Protein und 10 Gew.-% RNA.
Das gramnegative Bakterium Escherichia coli ist ein Modellsystem für die Nukleoidforschung, wie chromosomale DNA zum Nukleoid wird, welche Faktoren daran beteiligt sind, was über ihre Struktur bekannt ist und wie einige der DNA-Strukturaspekte die Genexpression beeinflussen .
Es gibt zwei wesentliche Aspekte der Nukleoidbildung; Kondensation einer großen DNA in einen kleinen Zellraum und funktionelle Organisation der DNA in einer dreidimensionalen Form. Das haploide zirkuläre Chromosom in E. coli besteht aus ~ 4,6 x 10 6 bp. Wenn DNA in der entspannten werden B - Form , würde es einen Umfang von ca. 1,5 Millimetern (0,332 nm x 4,6 x 10 6 ). Jedoch bleibt ein großes DNA-Molekül wie die chromosomale DNA von E. coli kein gerades starres Molekül in einer Suspension. Brownsche Bewegung erzeugt Krümmungen und Krümmungen in der DNA. Die maximale Länge, bis zu der eine doppelhelikale DNA durch Widerstand gegen die durch die Brownsche Bewegung erzwungene Biegung gerade bleibt, beträgt ~50 nm oder 150 bp, was als Persistenzlänge bezeichnet wird . Somit wird reine DNA ohne zusätzliche Faktoren im Wesentlichen kondensiert; im thermischen Gleichgewicht nimmt es eine zufällige Spulenform an . Die Zufallsknäuel von E. coli chromosomale DNA würde ein Volumen (4/3 π r besetzen 3 ) von ~ 523 & mgr; m 3 , berechnet aus dem Trägheitsradius ( R g = (√N a) / √6) wo a die ist Kuhn-Länge (2 x Persistenzlänge) und N ist die Anzahl der Kuhn-Längensegmente in der DNA (Gesamtlänge der DNA geteilt durch a ). Obwohl die DNA bereits in der zufälligen Spulenform kondensiert ist, kann sie immer noch nicht das Volumen des Nukleoids annehmen, das weniger als ein Mikrometer beträgt. Daher ist die inhärente Eigenschaft der DNA nicht ausreichend: zusätzliche Faktoren müssen dazu beitragen, die DNA weiter in der Größenordnung von ~10 3 (Volumen der Zufallsspirale geteilt durch das Nukleoidvolumen) zu kondensieren . Der zweite wesentliche Aspekt der Nukleoidbildung ist die funktionelle Anordnung der DNA. Chromosomale DNA ist nicht nur kondensiert, sondern auch funktionell so organisiert, dass sie mit DNA-Transaktionsprozessen wie Replikation , Rekombination , Segregation und Transkription kompatibel ist . Fast fünf Jahrzehnte Forschung seit 1971 haben gezeigt, dass die endgültige Form des Nukleoids aus einer hierarchischen Organisation der DNA entsteht. Im kleinsten Maßstab (1 kb oder weniger) kondensieren und organisieren nukleoid-assoziierte DNA-Architekturproteine DNA durch Biegen, Schleifen, Verbrücken oder Umhüllen von DNA. In einem größeren Maßstab (10 kb oder größer) bildet DNA plektonemische Schleifen, eine geflochtene Form von DNA, die durch Supercoiling induziert wird. Auf der Megabasenskala verschmelzen die plektonemischen Schleifen zu sechs räumlich organisierten Domänen (Makrodomänen), die durch häufigere physikalische Interaktionen zwischen DNA-Stellen innerhalb derselben Makrodomäne als zwischen verschiedenen Makrodomänen definiert werden. In und zwischen den Makrodomänen gebildete lang- und kurzreichweitige DNA-DNA-Verbindungen tragen zur Kondensation und funktionellen Organisation bei. Schließlich ist das Nukleoid ein helixförmiges Ellipsoid mit Regionen hochkondensierter DNA an der Längsachse.
Kondensation und Organisation
Nukleoid-assoziierte Proteine (NAPs)
In Eukaryoten ist genomische DNA in Form einer sich wiederholenden Anordnung von DNA-Protein-Partikeln, die als Nukleosomen bezeichnet werden, kondensiert .
Ein Nukleosom besteht aus ~146 bp DNA, die um einen octameren Komplex der Histonproteine gewickelt ist . Obwohl Bakterien keine Histone besitzen, besitzen sie eine Gruppe von DNA-bindenden Proteinen, die als Nukleoid-assoziierte Proteine (NAPs) bezeichnet werden und im weitesten Sinne funktionell analog zu Histone sind. NAPs sind sehr reichlich vorhanden und machen einen signifikanten Anteil der Proteinkomponente von Nukleoiden aus.
Ein charakteristisches Merkmal von NAPs ist ihre Fähigkeit, DNA sowohl spezifisch (entweder sequenz- oder strukturspezifisch) als auch nicht sequenzspezifisch zu binden. Als Ergebnis sind NAPs Proteine mit doppelter Funktion. Die spezifische Bindung von NAPs ist hauptsächlich an der genspezifischen Transkription , DNA-Replikation , Rekombination und Reparatur beteiligt . Auf dem Höhepunkt ihrer Häufigkeit ist die Zahl der Moleküle vieler NAPs um mehrere Größenordnungen höher als die Zahl der spezifischen Bindungsstellen im Genom. Daher wird vermutet, dass NAPs meist nicht sequenzspezifisch an die chromosomale DNA binden, und dieser Modus ist entscheidend für die Chromosomenverdichtung. Es ist bemerkenswert, dass die sogenannte nicht-sequenzspezifische Bindung eines NAP möglicherweise nicht vollständig zufällig ist. Aufgrund der sequenzabhängigen DNA-Konformation oder der von anderen NAPs erzeugten DNA-Konformation kann eine niedrige Sequenzspezifität und/oder strukturelle Spezifität vorliegen.
Obwohl die molekularen Mechanismen, wie NAPs kondensiert DNA in vivo nicht gut verstanden ist , auf der Basis der umfangreichen in vitro Studien scheint es , dass NAPs in Chromosoms Verdichtung über die folgenden Mechanismen teilnehmen: NAPs induzieren und stabilisieren Biegungen in DNA, was unterstützen DNA Kondensation durch Reduzierung die Persistenzlänge. NAPs kondensieren DNA durch Brückenbildung, Umhüllung und Bündelung, die zwischen benachbarten DNA-Segmenten oder entfernten DNA-Segmenten des Chromosoms auftreten können. Ein weiterer Mechanismus, durch den NAPs an der Chromosomenverdichtung teilnehmen, besteht darin, negative Supercoils in der DNA einzuschränken und so zur topologischen Organisation des Chromosoms beizutragen.
In E. coli wurden mindestens 12 NAPs identifiziert , von denen HU, IHF, H-NS und Fis am ausführlichsten untersucht wurden. Ihre Häufigkeit und DNA-Bindungseigenschaften und ihre Wirkung auf die DNA-Kondensation und -Organisation sind in den folgenden Tabellen zusammengefasst.
| Protein | Molekulargewicht (kDa) | Native Funktionseinheit | Abundanz 1 in der Wachstumsphase | Abundanz 1 in der stationären Phase |
|---|---|---|---|---|
| HUα und HUβ | ~ 9 | Homo- und Heterodimer | 55.000 (23) | 30.000 (12,5) |
| IHFα und IHFβ | ~ 11 | Heterodimer | 12.000 (5) | 55.000 (23) |
| H-NS | ~ 15 | Homodimer | 20.000 (8) | 15.000 (6) |
| Fis | ~ 11 | Homodimer | 60.000 (25) | Unauffindbar |
| Dps | ~ 19 | Dodecamer | 6.000 (0,4) | 180.000 (12,5) |
1 Daten zur Abundanz (Moleküle/Zelle) wurden entnommen; Die Zahl in Klammern ist die mikromolare Konzentration, die nach der folgenden Formel berechnet wird: (Zahl der nativen funktionellen Einheiten/Avogadro-Zahl) x (1/Zellvolumen in Liter) x 10 3 . Das Zellvolumen in Liter (2 x 10 –15 ) wurde bestimmt, indem ein Volumen der E. coli- Zelle von 2 μm 3 angenommen wurde .
| Protein | Bindungsmotiv | Spezifische DNA-Bindungsaffinität 1 | Zufällige DNA-Bindungsaffinität 1 |
|---|---|---|---|
| HU | Ein Strukturmotiv, das durch Biegungen und Knicke in der DNA definiert wird | 7,5 x 10 -9 | 4,0 x 10 -7 |
| H-NS | WATCAANNNNTTR | 1,5 x 10 -9 | 1,7 x 10 -6 |
| IHF | TCGATAAATT | 10-15 x 10 -9 | 6 x 10 -8 |
| Fis | GNTYAAAWTTTRANC | 0,2-1,0 x 10 -9 | >8,0 x 10 -6 |
| Dps | ND | ND | 1,65 x 10 -7 |
| MatP | GTGACRNYGTCAC | 8,0 x 10 -9 | ND |
| MukBEF | ND | ND | ND |
1 Bindungsaffinität bezieht sich auf die Gleichgewichtsdissoziationskonstante (Kd) in molaren Einheiten (M). ND = nicht bestimmt
HU
Das histonähnliche Protein aus dem E. coli- Stamm U93 (HU) ist ein evolutionär konserviertes Protein in Bakterien. HU existiert in E. coli als Homo- und Heterodimere von zwei Untereinheiten HUα und HUβ, die 69% Aminosäureidentität teilen. Obwohl es als ein Histon-ähnliches Protein bezeichnet wird, in der Nähe funktionelle Verwandten HU in Eukaryoten sind Hochmobilitätsgruppe (HMG) Proteine und nicht Histone. HU ist ein nicht sequenzspezifisches DNA-bindendes Protein. Es bindet mit geringer Affinität an jede lineare DNA. Es bindet jedoch bevorzugt mit hoher Affinität an eine strukturell verzerrte DNA. Beispiele für verzerrte DNA-Substrate umfassen kreuzförmige DNA , ausgebauchte DNA, dsDNA, die einen Einzelstrangbruch wie Nicks , Gaps oder Forks enthält . Darüber hinaus bindet und stabilisiert HU spezifisch eine Protein-vermittelte DNA-Schleife. Im strukturspezifischen DNA-Bindungsmodus erkennt HU ein gemeinsames Strukturmotiv, das durch durch Verzerrung erzeugte Biegungen oder Knicke definiert ist, während es an eine lineare DNA bindet, indem es das Phosphatrückgrat verriegelt. Während die strukturspezifische Bindung mit hoher Affinität für spezialisierte Funktionen von HU wie die ortsspezifische Rekombination , DNA-Reparatur , DNA-Replikationsinitiation und Genregulation erforderlich ist , scheint die allgemeine Bindung mit niedriger Affinität an der DNA-Kondensation beteiligt zu sein. Bei Chromatin-Immunpräzipitation gekoppelt mit DNA-Sequenzierung ( ChIP-Seq ) zeigt HU keine spezifischen Bindungsereignisse. Stattdessen zeigt es eine einheitliche Bindung über das Genom, die vermutlich seine meist schwache, nicht sequenzspezifische Bindung widerspiegelt, wodurch die hochaffine Bindung in vivo maskiert wird .
In Stämmen, denen HU fehlt, wird das Nukleoid "dekondensiert", was mit einer Rolle von HU bei der DNA-Kompaktierung übereinstimmt. Die folgenden in vitro - Studien legen mögliche Mechanismen nahe , wie HU DNA in vivo kondensieren und organisieren könnte . HU bindet nicht nur stabil an verzerrte DNA mit Biegungen, es induziert auch in einer linearen DNA bei einer Konzentration von weniger als 100 nM flexible Biegungen. Im Gegensatz dazu zeigt HU bei höheren physiologisch relevanten Konzentrationen den gegenteiligen Architektureffekt auf die DNA. Es bildet starre Nukleoprotein-Filamente, die das Straitening der DNA und nicht die Biegung verursachen. Die Filamente können außerdem aufgrund der durch die nicht sequenzspezifische DNA-Bindung ausgelösten HU-HU-Multimerisierung ein sowohl lateral als auch medial expandierbares DNA-Netzwerk (DNA-Bunching) bilden.
Inwiefern sind diese Verhaltensweisen von HU innerhalb der Zelle relevant? Die Bildung von Filamenten erfordert eine hochdichte Bindung von HU an DNA, ein HU-Dimer pro 9-20 bp DNA. Aber es gibt nur ein HU-Dimer alle ~150 bp der chromosomalen DNA, basierend auf der geschätzten Häufigkeit von 30.000 HU-Dimeren pro Zelle (4600000 bp /30.000). Dies weist darauf hin, dass die flexiblen Biegungen wahrscheinlicher in vivo auftreten . Die flexible Biegung würde eine Kondensation aufgrund einer Verringerung der Persistenzlänge der DNA verursachen, wie durch magnetische Pinzettenexperimente gezeigt , die es ermöglichen, die Kondensation eines einzelnen DNA-Moleküls durch ein DNA-bindendes Protein zu untersuchen. Aufgrund der Kooperativität könnten sich jedoch in einigen Regionen des Chromosoms die starren Filamente und Netzwerke bilden. Die Filamentbildung allein induziert keine Kondensation, aber DNA-Netzwerke oder -Bündelung können wesentlich zur Kondensation beitragen, indem sie entfernte oder nahegelegene Chromosomensegmente zusammenbringen.
IHF
Integration Host Factor (IHF) ist strukturell fast identisch mit HU, verhält sich aber in vielen Aspekten anders als HU. Im Gegensatz zu HU, das unabhängig von der Sequenz bevorzugt an ein Strukturmotiv bindet, bindet IHF bevorzugt an eine spezifische DNA-Sequenz, obwohl die Spezifität durch die sequenzabhängige DNA-Struktur und Verformbarkeit entsteht. Die spezifische Bindung von IHF an verwandten Stellen verbiegt die DNA stark um >160 Grad. Das verwandte Sequenzmotiv kommt im Genom von E. coli bei etwa 3000 vor . Die geschätzte Häufigkeit von IHF in der Wachstumsphase beträgt etwa 6000 Dimere pro Zelle. Unter der Annahme, dass ein IHF-Dimer an ein einzelnes Motiv bindet und das Nukleoid während der exponentiellen Wachstumsphase mehr als ein Genomäquivalent enthält, würden die meisten IHF-Moleküle spezifische Stellen im Genom besetzen und DNA wahrscheinlich nur durch eine scharfe Krümmung kondensieren.
Neben der bevorzugten Bindung an eine spezifische DNA-Sequenz bindet IHF auch nicht sequenzspezifisch an DNA mit ähnlichen Affinitäten wie HU. Eine Rolle der unspezifischen Bindung von IHF bei der DNA-Kondensation scheint in der stationären Phase kritisch zu sein, da die IHF-Häufigkeit in der stationären Phase um das Fünffache ansteigt und die zusätzlichen IHF-Dimere wahrscheinlich unspezifisch an die chromosomale DNA binden würden. Im Gegensatz zu HU bildet IHF bei höheren Konzentrationen keine dicken starren Filamente. Stattdessen induziert seine unspezifische Bindung auch eine DNA-Biegung, obwohl der Grad der Biegung viel geringer ist als der an spezifischen Stellen und ähnlich der flexiblen Biegung, die durch HU in einer linearen DNA bei niedrigen Konzentrationen induziert wird. In vitro kann die durch unspezifische Bindung von IHF induzierte Biegung eine DNA-Kondensation verursachen und die Bildung von Nukleoproteinkomplexen höherer Ordnung in Abhängigkeit von den Konzentrationen von Kaliumchlorid und Magnesiumchlorid fördern. Die DNA-Organisation höherer Ordnung durch IHF in vivo ist noch unklar.
H-NS
Ein unterscheidbares Merkmal des histonähnlichen oder hitzestabilen nukleoiden Strukturierungsproteins (H-NS) von anderen NAPs ist die Fähigkeit, bei relativ niedrigen Konzentrationen (<1 x 10 −5 M) von der homodimeren Form in einen oligomeren Zustand bei höheren zu wechseln Ebenen. Aufgrund der Oligomerisierungseigenschaften breitet sich H-NS in einer Nukleationsreaktion seitlich entlang der AT-reichen DNA aus , wobei hochaffine Stellen als Nukleationszentren fungieren. Die Ausbreitung von H-NS auf DNA führt in Abhängigkeit von der Magnesiumkonzentration in der Reaktion zu zwei gegensätzlichen Ergebnissen. Bei niedriger Magnesiumkonzentration (< 2 mM) bildet H-NS starre Nukleoproteinfilamente, während es bei höheren Magnesiumkonzentrationen (> 5 mM) inter- und intramolekulare Brücken bildet. Die Bildung von starren Filamenten führt zu einer Begradigung der DNA ohne Kondensation, während die Überbrückung eine erhebliche DNA-Faltung verursacht. Die Analyse der H-NS-Bindung im Genom durch ChIP-Seq- Assays lieferte einen indirekten Beweis für die Ausbreitung von H-NS auf DNA in vivo . H-NS bindet selektiv an 458 Regionen im Genom. Obwohl gezeigt wurde, dass H-NS gekrümmte DNA bevorzugt, die durch wiederholte A-Spuren in DNA-Sequenzen gebildet wird, ist die Grundlage der selektiven Bindung das Vorhandensein eines konservierten Sequenzmotivs, das in AT-reichen Regionen gefunden wird. Noch wichtiger ist, dass das häufige Auftreten des Sequenzmotivs innerhalb einer H-NS-Bindungsregion, das die kooperativen Protein-Protein-Wechselwirkungen verstärken kann, und die ungewöhnlich lange Länge der Bindungsregion mit der Ausbreitung des Proteins vereinbar sind. Ob die Filamentbildung oder die DNA-Brückenbildung in vivo vorherrscht, hängt von der physiologischen Magnesiumkonzentration innerhalb der Zelle ab. Wenn die Magnesiumkonzentration gleichmäßig niedrig ist (< 5 mM), würde H-NS in vivo starre Nukleoproteinfilamente bilden . Alternativ, wenn es eine ungleichmäßige Verteilung von Magnesium in der Zelle gibt, könnte es sowohl die DNA-Brücke als auch die Versteifung fördern, jedoch in verschiedenen Regionen des Nukleoids.
Darüber hinaus ist H-NS am besten als globaler Gen-Silencer bekannt, der vorzugsweise die Transkription von horizontal übertragenen Genen hemmt, und es ist das starre Filament, das zum Gen-Silencing führt. Zusammengefasst scheint es, dass die Bildung von starren Filamenten das wahrscheinlichste Ergebnis von H-NS-DNA-Wechselwirkungen in vivo ist , die zu Gen-Silencing führen, aber keine DNA-Kondensation induzieren. Konsequenterweise verändert die Abwesenheit von H-NS das Nukleoidvolumen nicht. Es ist jedoch möglich, dass E. coli unter bestimmten Umgebungsbedingungen eine hohe Magnesiumkonzentration aufweist. Unter solchen Bedingungen kann H-NS von seiner filamentinduzierenden Form in die brückeninduzierende Form wechseln, die zur DNA-Kondensation und -Organisation beiträgt.
Fis
Faktor für die Inversionsstimulation (Fis) ist ein sequenzspezifisches DNA-bindendes Protein, das an spezifische DNA-Sequenzen bindet, die ein symmetrisches 15-bp-Motiv enthalten. Wie IHF induziert Fis die DNA-Biegung an verwandten Stellen. Die Fähigkeit, DNA zu biegen, zeigt sich in der Struktur des Fis-Homodimers. Ein Fis-Homodimer besitzt zwei Helix-Turn-Helix (HTH)-Motive, eines von jedem Monomer. Ein HTH-Motiv erkennt typischerweise die DNA-Hauptfurche. Der Abstand zwischen den DNA-Erkennungshelices der beiden HTH-Motive im Fis-Homodimer ist jedoch 25 Å , also ~ 8 Å kürzer als die Ganghöhe einer kanonischen B-DNA , was darauf hindeutet, dass das Protein DNA biegen oder verdrehen muss, um stabil zu binden . Konsequenterweise zeigt die Kristallstruktur von Fis-DNA-Komplexen, dass der Abstand zwischen den Erkennungshelices unverändert bleibt, während die DNA-Kurven im Bereich von 60-75 Grad liegen. Es gibt 1464 Fis-Bindungsregionen, die über das Genom von E. coli verteilt sind, und ein rechnerisch identifiziertes Bindungsmotiv stimmt mit dem bekannten 15-bp-Motiv überein. Eine spezifische Bindung von Fis an solchen Stellen würde Krümmungen in der DNA induzieren und somit zur DNA-Kondensation beitragen, indem die Persistenzlänge der DNA verringert wird. Darüber hinaus treten viele Fis-Bindungsstellen hintereinander auf, wie die in den stabilen RNA-Promotoren, zB dem P1- Promotor des rRNA- Operons rrnB . Die kohärente Biegung durch Fis an den Tandemstellen erzeugt wahrscheinlich eine DNA-Mikroschleife, die weiter zur DNA-Kondensation beitragen kann.
Neben einer hochaffinen spezifischen Bindung an verwandte Stellen kann Fis an eine zufällige DNA-Sequenz binden. Die unspezifische DNA-Bindung ist signifikant, da Fis in der Wachstumsphase so häufig vorkommt wie HU . Daher wird erwartet, dass die meisten Fis-Moleküle DNA nicht sequenzspezifisch binden. Experimente mit magnetischen Pinzetten zeigen, dass diese unspezifische Bindung von Fis zur DNA-Kondensation und -Organisation beitragen kann. Fis verursacht eine leichte Kondensation eines einzelnen DNA-Moleküls bei <1 mM, induziert jedoch eine erhebliche Faltung durch die Bildung von DNA-Schleifen mit einer durchschnittlichen Größe von ~800 bp bei >1 mM. Die Schleifen in magnetischen Pinzetten-Experimenten unterscheiden sich von den Mikroschleifen, die durch kohärente DNA-Biegung an verwandten Stellen erzeugt werden, da sie die Bildung von DNA-Protein-Komplexen hoher Dichte erfordern, die durch sequenzunabhängige Bindung erreicht werden. Obwohl das Auftreten solcher Schleifen in vivo noch gezeigt werden muss, kann eine hochdichte Bindung von Fis in vivo durch eine konzertierte Wirkung sowohl der spezifischen als auch der unspezifischen Bindung erfolgen. Das gleichzeitige Auftreten spezifischer Stellen könnte eine Nukleationsreaktion ähnlich der von H-NS auslösen, und dann würde eine unspezifische Bindung zur Bildung von lokalisierten hochdichten Fis-Arrays führen. Die Überbrückung zwischen diesen lokalisierten Regionen kann große DNA-Schleifen erzeugen. Fis kommt ausschließlich in der Wachstumsphase und nicht in der stationären Phase vor . Somit muss jede Rolle bei der chromosomalen Kondensation durch Fis spezifisch für wachsende Zellen sein.
Nukleoid-assoziierte RNAs (naRNAs)
Frühe Studien, die die Wirkung der RNase A-Behandlung auf isolierte Nukleoide untersuchten, zeigten, dass RNA an der Stabilisierung des Nukleoids im kondensierten Zustand beteiligt war. Darüber hinaus zerbrach die Behandlung mit RNase A die DNA-Fasern in dünnere Fasern, wie durch eine Rasterkraftmikroskopie des Nukleoids unter Verwendung des „On-Substrat-Lyse-Verfahrens“ beobachtet wurde. Diese Ergebnisse zeigten die Beteiligung von RNA an der Nukleoidstruktur, aber die Identität des RNA-Moleküls bzw. der RNA-Moleküle blieb bis vor kurzem unbekannt. Die meisten Studien zu HU konzentrierten sich auf seine DNA-Bindung. HU bindet jedoch auch an dsRNA- und RNA-DNA-Hybride mit einer geringeren Affinität ähnlich der einer linearen dsDNA. Darüber hinaus bindet HU bevorzugt an RNA, die Sekundärstrukturen enthält, und an ein RNA-DNA-Hybrid, in dem die RNA einen Nick oder Überhang enthält. Die Bindungsaffinitäten von HU zu diesen RNA-Substraten sind denen ähnlich, mit denen es an verzerrte DNA bindet. Eine Immunpräzipitation von HU-gebundener RNA gekoppelt an eine Reverse Transkription und Microarray (RIP-Chip) Studie sowie eine Analyse von RNA aus gereinigten intakten Nukleoiden identifizierten Nukleoid-assoziierte RNA-Moleküle, die mit HU interagieren. Mehrere von ihnen sind nicht-kodierende RNAs, und eine solche RNA namens naRNA4 (Nukleoid-assoziierte RNA 4) wird in einem repetitiven extragenen Palindrom ( REP325 ) kodiert . In einem Stamm ohne REP325 wird das Nukleoid dekondensiert wie in einem Stamm ohne HU. naRNA4 nimmt höchstwahrscheinlich an der DNA-Kondensation teil, indem es DNA-Segmente in Gegenwart von HU verbindet. Neuere Studien liefern Einblicke in den molekularen Mechanismus, wie naRNA4 DNA-DNA-Verbindungen herstellt. Die RNA zielt auf DNA-Regionen ab, die kreuzförmige Strukturen enthalten, und bildet einen RNA-DNA-Komplex, der für die Herstellung von DNA-DNA-Verbindungen entscheidend ist. Obwohl HU bei der Bildung des Komplexes hilft, ist es überraschenderweise nicht im endgültigen Komplex vorhanden, was auf seine potenzielle Rolle als Katalysator (Chaperon) hinweist. Die Natur des RNA-DNA-Komplexes bleibt rätselhaft, da die Bildung des Komplexes keine ausgedehnte Watson/Crick-Basenpaarung beinhaltet, sondern empfindlich gegenüber RNase H ist, die RNA in einem RNA-DNA-Hybrid spaltet und der Komplex an einen Antikörper bindet, der für RNA-DNA-Hybride.
Supercoiling
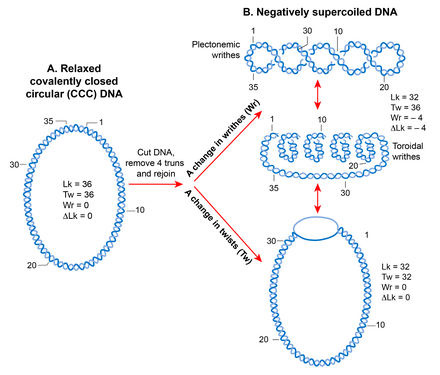
Aufgrund seiner helikalen Struktur wird ein doppelsträngiges DNA-Molekül topologisch in die kovalent geschlossene zirkuläre Form eingeschränkt, wodurch die Rotation der freien Enden eliminiert wird. Die Häufigkeit, mit der sich die beiden Stränge in einer topologisch eingeschränkten DNA kreuzen, wird als Verknüpfungszahl (Lk) bezeichnet, die der Anzahl der helikalen Windungen oder Verdrillungen in einem kreisförmigen Molekül entspricht. Die Lk einer topologischen DNA bleibt invariant, egal wie das DNA-Molekül verformt wird, solange kein Strang gebrochen ist.
Der Lk der DNA in der entspannten Form wird als Lk 0 definiert . Für jede DNA kann Lk 0 berechnet werden, indem die Länge (in bp) der DNA durch die Anzahl von bp pro Helixwindung geteilt wird. Dies entspricht 10,4 bp für die relaxierte B-Form-DNA . Jede Abweichung von Lk 0 verursacht Supercoiling in der DNA. Eine Verringerung der Verknüpfungszahl (Lk < Lk 0 ) erzeugt eine negative Supercoiling, während eine Erhöhung der Verknüpfungszahl (Lk > Lk 0 ) eine positive Supercoiling erzeugt.
Der supercoiled-Zustand (wenn Lk nicht gleich Lk 0 ist ) führt zu einem Übergang in der DNA-Struktur, der sich als Änderung der Anzahl der Drehungen (negativ < 10,4 bp/Umdrehung, positiv > 10,4 bp/Umdrehung) und/oder in . manifestieren kann die Bildung von Krümmungen , sogenannte Supercoils. Somit ist Lk mathematisch als eine vorzeichenabhängige Summe der beiden geometrischen Parameter Twist und Writhe definiert. Ein quantitatives Maß für das Supercoiling, das unabhängig von der Größe der DNA-Moleküle ist, ist die Supercoiling-Dichte (σ) mit = ∆Lk/Lk 0 .
Krümmungen können zwei Strukturen annehmen; Plektonem und Solenoid oder Toroid. Durch die Verwindung der Helixachse entsteht eine plektonemische Struktur. Toroide Superspulen entstehen, wenn die DNA mehrere Spiralen um eine Achse bildet und sich nicht kreuzt, wie bei einem Telefonkabel. Die Krümmungen in der Plektonemform sind in positiv bzw. negativ supercoiled DNA rechts- und linkshändig. Die Händigkeit der toroidalen Superspulen ist der von Plektonemen entgegengesetzt. Sowohl Plektoneme als auch toroidale Supercoils können entweder in freier Form vorliegen oder in gebundener Form mit Proteinen zurückgehalten werden. Das beste Beispiel für das gebundene toroidale Supercoiling in der Biologie ist das eukaryotische Nukleosom, in dem sich DNA um Histone wickelt .
Plektonemische Superspulen in E. coli

In den meisten Bakterien liegt die DNA in supercoiled Form vor. Die zirkuläre Natur des E. coli- Chromosoms macht es zu einem topologisch eingeschränkten Molekül, das hauptsächlich negativ supercoiled ist mit einer geschätzten durchschnittlichen Supercoiling-Dichte (σ) von –0,05. Im eukaryontischen Chromatin findet sich DNA hauptsächlich in der toroidalen Form, die durch die Bildung von Nukleosomen durch Histone zurückgehalten und definiert wird. Im Gegensatz dazu ist im Nukleoid von E. coli etwa die Hälfte der chromosomalen DNA in Form von freien, plektonemischen Supercoils organisiert. Die verbleibende DNA wird durch Wechselwirkung mit Proteinen wie NAPs entweder in der plektonämischen Form oder in alternativen Formen, einschließlich, aber nicht beschränkt auf die toroidale Form, zurückgehalten. Somit stellen plektonemische Supercoils ein effektives Supercoiling des E. coli- Genoms dar, das für seine Kondensation und Organisation verantwortlich ist. Sowohl das plektonemische als auch das toroidale Supercoiling unterstützen die DNA-Kondensation. Es ist bemerkenswert, dass es aufgrund der Verzweigung der plektonemischen Strukturen weniger DNA-Kondensation liefert als die toroidale Struktur. Ein DNA-Molekül gleicher Größe mit gleichen Supercoiling-Dichten ist in einer toroidalen Form kompakter als in einer plektonemischen Form. Neben der DNA-Kondensation hilft Supercoiling bei der DNA-Organisation. Es fördert die Entflechtung der DNA, indem es die Wahrscheinlichkeit einer Verkettung verringert. Supercoiling hilft auch, zwei entfernte DNA-Stellen in die Nähe zu bringen, wodurch eine potenzielle funktionelle Wechselwirkung zwischen verschiedenen DNA-Segmenten gefördert wird.
Quellen des Supercoilings in E. coli
Drei Faktoren tragen zur Erzeugung und Aufrechterhaltung des chromosomalen DNA-Supercoiling in E. coli bei : (i) Aktivitäten von Topoisomerasen , (ii) der Transkriptionsvorgang und (iii) NAPs.
Topoisomerasen
Topoisomerasen sind eine besondere Kategorie von DNA-Stoffwechselenzymen, die Supercoiling erzeugen oder entfernen, indem sie DNA-Stränge brechen und dann erneut ligieren. E. coli besitzt vier Topoisomerasen. DNA-Gyrase führt in Gegenwart von ATP negatives Supercoiling ein und entfernt positives Supercoiling in Abwesenheit von ATP. In allen Lebensformen ist DNA-Gyrase die einzige Topoisomerase, die negative Supercoiling erzeugen kann, und aufgrund dieser einzigartigen Fähigkeit besitzen bakterielle Genome freie negative Supercoils; DNA-Gyrase kommt in allen Bakterien vor, fehlt jedoch bei höheren Eukaryoten. Im Gegensatz dazu wirkt Topo I der DNA-Gyrase entgegen, indem es die negativ supercoiled DNA relaxiert. Es gibt genetische Beweise dafür, dass ein Gleichgewicht zwischen den gegensätzlichen Aktivitäten von DNA-Gyrase und Topo I für die Aufrechterhaltung eines stationären Niveaus der durchschnittlichen negativen Superhelizität in E. coli verantwortlich ist . Beide Enzyme sind für das Überleben von E. coli essentiell . Ein Null-Stamm von topA , dem Gen, das für Topo I kodiert, überlebt nur wegen der Anwesenheit von Suppressor-Mutationen in den Genen, die für DNA-Gyrase kodieren. Diese Mutationen führen zu einer verringerten Gyrase-Aktivität, was darauf hindeutet, dass ein übermäßiges negatives Supercoiling aufgrund des Fehlens von Topo I durch eine verringerte negative Supercoiling-Aktivität der DNA-Gyrase kompensiert wird. Topo III ist in E. coli entbehrlich und es ist nicht bekannt, dass es beim Supercoiling in E. coli eine Rolle spielt . Die Hauptfunktion von Topo IV besteht darin, Schwesterchromosomen aufzulösen. Es hat sich jedoch gezeigt, dass es auch zum stationären Niveau des negativen Supercoilings beiträgt, indem es das negative Supercoiling zusammen mit Topo I entspannt.
| Topoisomerase | Typ | Funktion | Einzel- oder doppelsträngige Spaltung |
|---|---|---|---|
| Topoisomerase I | NS | Entfernt (-) Supercoiling | SS |
| Topoisomerase III | NS | Entfernt (-) Supercoiling | SS |
| Topoisomerase IV | IIA | Entfernt (-) Supercoiling | DS |
| DNA-Gyrase | IIA | Erzeugt (-) Supercoiling und entfernt (+) Supercoiling | DS |
Transkription
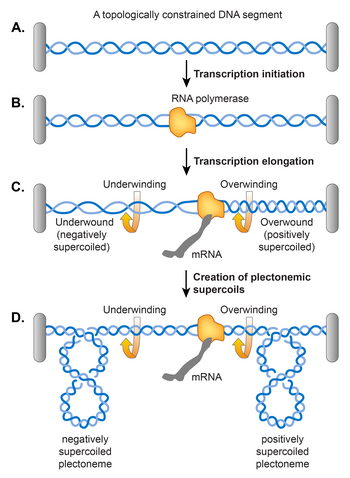
Ein von Liu und Wang vorgeschlagenes Zwillings-Supercoiling-Domänenmodell argumentierte, dass die Entwindung der DNA-Doppelhelix während der Transkription ein Supercoiling in der DNA induziert, wie in gezeigt. Gemäß ihrem Modell zwingt die transkribierende RNA-Polymerase (RNAP), die entlang der DNA gleitet, die DNA dazu, sich um ihre helikale Achse zu drehen . Eine Behinderung der freien DNA-Rotation könnte aufgrund einer topologischen Einschränkung auftreten, die dazu führt, dass die DNA vor der RNAP überdreht (positiv supercoiled) und die DNA hinter der RNAP unterdreht (negativ supercoiled) wird. Es wurde festgestellt, dass eine topologische Einschränkung nicht erforderlich ist, da RNAP ein ausreichendes Drehmoment erzeugt, das sogar in einer linearen DNA-Matrize ein Supercoiling verursacht. Wenn DNA bereits negativ supercoiled ist, entspannt diese Aktion vorhandene negative Supercoils, bevor eine Ansammlung positiver Supercoils vor RNAP verursacht wird, und führt mehr negative Supercoils hinter RNAP ein. Im Prinzip sollten DNA-Gyrase und Topo I überschüssige positive bzw. negative Supercoils entfernen, aber wenn die RNAP-Elongationsrate den Umsatz der beiden Enzyme überschreitet, trägt die Transkription zum stabilen Supercoiling-Niveau bei.
Kontrolle des Supercoilings durch NAPs
Im eukaryotischen Chromatin ist DNA selten in der freien Supercoiling-Form vorhanden, da Nukleosomen fast alle negativen Supercoilings durch enge Bindung der DNA an Histone hemmen. In ähnlicher Weise beschränken in E. coli Nukleoproteinkomplexe, die durch NAPs gebildet werden, die Hälfte der Supercoiling-Dichte des Nukleoids. Mit anderen Worten, wenn ein NAP von einem Nukleoproteinkomplex dissoziiert , würde die DNA die freie, plektonemische Form annehmen. Es wurde experimentell gezeigt, dass die DNA-Bindung von HU, Fis und H-NS negatives Supercoiling in einer entspannten, aber topologisch eingeschränkten DNA hemmt. Sie können dies tun, indem sie entweder die helikale Ganghöhe der DNA ändern oder toroidale Krümmungen durch Biegen und Umhüllen der DNA erzeugen. Alternativ können NAPs bevorzugt an andere Formen der unterwundenen DNA wie kreuzförmige Strukturen und verzweigte Plektoneme binden und diese stabilisieren. Es wurde berichtet, dass Fis verzweigte Plektoneme durch seine Bindung an Cross-Over-Regionen organisiert und HU bevorzugt an kreuzförmige Strukturen bindet.
NAPs regulieren auch indirekt das DNA-Supercoiling. Fis kann Supercoiling modulieren, indem es die Transkription der Gene, die für DNA-Gyrase kodieren, unterdrückt. Es gibt genetische Beweise dafür, dass HU die Supercoiling-Spiegel durch Stimulierung der DNA-Gyrase und Verringerung der Aktivität von Topo I kontrolliert. Zur Unterstützung der genetischen Studien wurde gezeigt, dass HU die DNA-Gyrase-katalysierte Dekatenation von DNA in vitro stimuliert . Es ist mechanistisch unklar, wie HU die Aktivitäten der Gyrase und Topo I moduliert. HU könnte physikalisch mit DNA-Gyrase und Topo I interagieren oder DNA-Organisationsaktivitäten von HU wie DNA-Biegung können die Wirkung von DNA-Gyrase bzw. Topo I erleichtern oder hemmen.
Plektonemische Superspulen gliedern sich in mehrere topologische Domänen
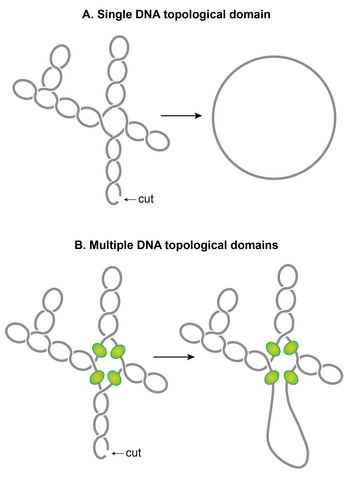
Eines der bemerkenswerten Merkmale des Nukleoids ist, dass plektonemische Superspulen in mehrere topologische Domänen organisiert sind. Mit anderen Worten, ein einzelner Schnitt in einer Domäne entspannt nur diese Domäne und nicht die anderen. Eine topologische Domäne bildet sich aufgrund einer Supercoiling-Diffusions-Barriere. Unabhängige Studien mit unterschiedlichen Methoden haben berichtet, dass die topologischen Domänen variabel in der Größe von 10–400 kb sind. Eine in diesen Studien häufig beobachtete zufällige Anordnung von Barrieren scheint die große Variabilität in der Größe der Domänen zu erklären.
Obwohl die Identitäten von Domänenbarrieren noch festgelegt werden müssen, sind mögliche Mechanismen, die für die Bildung der Barrieren verantwortlich sind, unter anderem: (i) Eine Domänenbarriere könnte sich bilden, wenn ein Protein mit der Fähigkeit, Supercoils zurückzuhalten, gleichzeitig an zwei unterschiedliche Stellen auf dem Chromosom bindet und topologisch isolierte DNA-Schleife oder -Domäne. Es wurde experimentell gezeigt, dass Protein-vermitteltes Looping in supercoiled DNA eine topologische Domäne erzeugen kann. NAPs wie H-NS und Fis sind aufgrund ihrer DNA-Looping-Fähigkeiten und der Verteilung ihrer Bindungsstellen potenzielle Kandidaten. (ii) Bakteriell durchsetzte Mosaikelemente (BIMEs) erscheinen ebenfalls als potenzielle Kandidaten für Domänenbarrieren. BIMEs sind palindromische Wiederholungssequenzen, die normalerweise zwischen Genen gefunden werden. Es wurde gezeigt, dass ein BIME die Diffusion von Supercoiling in einer synthetisch entworfenen topologischen Kassette, die in das E. coli- Chromosom eingefügt ist, behindert . Es gibt ~600 BIMEs, die über das Genom verteilt sind und das Chromosom möglicherweise in 600 topologische Domänen unterteilen. (iii) Barrieren könnten auch aus der Anlagerung von DNA an die Zellmembran durch ein Protein resultieren, das sowohl an DNA als auch an die Membran bindet, oder durch entstehende Transkription und die Translation von membranverankerten Proteinen. (iv) Transkriptionsaktivität kann Supercoiling-Diffusionsbarrieren erzeugen. Es wurde gezeigt, dass eine aktiv transkribierende RNAP die Dissipation von plektonemischen Supercoils blockiert und dadurch eine Supercoiling-Diffusions-Barriere bildet.
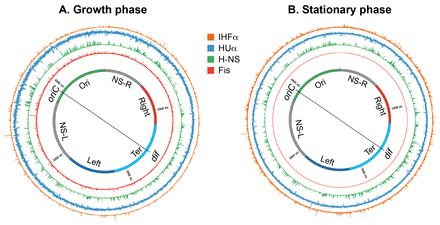
Wachstumsphasenabhängige Nukleoiddynamik
Das Nukleoid reorganisiert sich in Zellen der stationären Phase, was darauf hindeutet, dass die Nukleoidstruktur hochdynamisch ist, bestimmt durch den physiologischen Zustand der Zellen. Ein Vergleich von hochauflösenden Kontaktkarten des Nukleoids zeigte, dass die Fernkontakte in der Ter-Makrodomäne in der stationären Phase im Vergleich zur Wachstumsphase zunahmen . Darüber hinaus waren die CID-Grenzen in der stationären Phase anders als in der Wachstumsphase. Schließlich erfährt die nukleoide Morphologie während einer verlängerten stationären Phase eine massive Transformation; das Nukleoid weist geordnete, toroidale Strukturen auf.
Wachstumsphasenspezifische Veränderungen der Nukleoidstruktur könnten durch eine Veränderung der Konzentrationen von Nukleoid-assoziierten DNA-Architekturproteinen (den NAPs und der Muk-Untereinheiten), Supercoiling und Transkriptionsaktivität bewirkt werden. Die Häufigkeit der NAPs und der Muk-Untereinheiten ändert sich entsprechend dem bakteriellen Wachstumszyklus. Fis und das durch Hunger induzierte DNA-bindende Protein Dps, ein weiterer NAP, sind fast ausschließlich in der Wachstumsphase bzw. in der stationären Phase vorhanden. Die Fis-Spiegel steigen beim Eintritt in die exponentielle Phase an und fallen dann schnell ab, während sich die Zellen noch in der exponentiellen Phase befinden, und erreichen Werte, die in der stationären Phase nicht nachweisbar sind. Während die Fis-Werte zu sinken beginnen, beginnen die Dps-Werte zu steigen und erreichen in der stationären Phase ein Maximum. Ein dramatischer Übergang in der Nukleoidstruktur, der in der verlängerten stationären Phase beobachtet wurde, wurde hauptsächlich Dps zugeschrieben. Es bildet DNA/ kristalline Anordnungen, die das Nukleoid vor DNA-schädigenden Mitteln schützen, die während des Hungers vorhanden sind.
HU, IHF und H-NS sind sowohl in der Wachstumsphase als auch in der stationären Phase vorhanden. Ihre Häufigkeit ändert sich jedoch signifikant, sodass HU und Fis die häufigsten NAPs in der Wachstumsphase sind, während IHF und Dps die häufigsten NAPs in der stationären Phase werden. HUαα ist die vorherrschende Form in der frühen exponentiellen Phase, während die heterodimere Form in der stationären Phase mit geringen Mengen an Homodimeren vorherrscht. Dieser Übergang hat funktionelle Konsequenzen in Bezug auf die Nukleoidstruktur, da die beiden Formen die DNA anscheinend unterschiedlich organisieren und kondensieren; sowohl Homo- als auch Heterodimere bilden Filamente, aber nur das Homodimer kann mehrere DNA-Segmente zu einem DNA-Netzwerk zusammenbringen. Die Kopienzahl von MukB erhöht sich in der stationären Phase um das Zweifache. Eine Erhöhung der Anzahl von MukB-Molekülen könnte Einfluss auf die Prozessivität des MukBEF-Komplexes als einen DNA-Schleifen-Extrusionsfaktor haben, was zu einer größeren oder größeren Anzahl der Schleifen führt.
Supercoiling kann konzertiert mit DNA-Architekturproteinen wirken, um das Nukleoid zu reorganisieren. Das Gesamtniveau des Supercoilings nimmt in der stationären Phase ab, und das Supercoiling zeigt auf regionaler Ebene ein anderes Muster. Änderungen des Supercoilings können die topologische Organisation des Nukleoids verändern. Da außerdem eine chromosomale Region mit hoher Transkriptionsaktivität eine CID-Grenze bildet, könnten Änderungen der Transkriptionsaktivität während verschiedener Wachstumsphasen die Bildung von CID-Grenzen und damit die räumliche Organisation des Nukleoids verändern. Es ist möglich, dass in der stationären Phase beobachtete Veränderungen der CID-Grenzen auf die hohe Expression eines anderen Gensatzes in der stationären Phase im Vergleich zur Wachstumsphase zurückzuführen sind.
Nukleoidstruktur und Genexpression
NAPs und Genexpression
Die Chromosomenstruktur und Genexpression von E. coli scheinen sich gegenseitig zu beeinflussen. Einerseits weist eine Korrelation einer CID-Grenze mit hoher Transkriptionsaktivität darauf hin, dass die Chromosomenorganisation durch die Transkription getrieben wird. Andererseits kann die 3D-Struktur der DNA innerhalb des Nukleoids in jeder Größenordnung mit der Genexpression in Verbindung gebracht werden. Zunächst wurde gezeigt, dass die Reorganisation der 3D-Architektur des Nukleoids in E. coli das zelluläre Transkriptionsmuster dynamisch modulieren kann. Eine Mutante von HUa machte das Nukleoid durch eine erhöhte positive Superhelizität der chromosomalen DNA stark kondensiert. Folglich wurden viele Gene reprimiert und viele ruhende Gene wurden exprimiert. Außerdem gibt es viele spezifische Fälle, in denen proteinvermittelte lokale Architekturänderungen die Gentranskription verändern. Beispielsweise blockiert die Bildung von starren Nukleoproteinfilamenten durch H-NS den RNAP-Zugang zum Promotor und verhindert so die Gentranskription. Durch Gen-Silencing wirkt H-NS als globaler Repressor, der vorzugsweise die Transkription horizontal übertragener Gene hemmt. In einem anderen Beispiel erleichtert die spezifische Bindung von HU an das gal- Operon die Bildung einer DNA-Schleife, die das gal- Operon in Abwesenheit des Induktors reprimiert hält . Die topologisch unterschiedliche DNA-Mikroschleife, die durch kohärentes Biegen von DNA durch Fis an stabilen RNA-Promotoren entsteht, aktiviert die Transkription. DNA-Biegung durch IHF kontrolliert differenziell die Transkription von den beiden Tandem-Promotoren des ilvGMEDA- Operons in E. coli . Spezifische topologische Veränderungen durch NAPs regulieren nicht nur die Gentranskription, sondern sind auch an anderen Prozessen wie der DNA-Replikationsinitiation, Rekombination und Transposition beteiligt. Im Gegensatz zur spezifischen Genregulation muss noch untersucht werden, wie die Chromosomenstruktur höherer Ordnung und ihre Dynamik die Genexpression global auf molekularer Ebene beeinflusst.
DNA-Supercoiling und Genexpression
Zwischen DNA-Supercoiling und Gentranskription besteht eine wechselseitige Verbindung. Negatives Supercoiling der Promotorregion kann die Transkription stimulieren, indem das Schmelzen des Promotors erleichtert und die DNA-Bindungsaffinität eines Proteinregulators erhöht wird. Stochastische Bursts der Transkription scheinen ein allgemeines Merkmal von stark exprimierten Genen zu sein, und Supercoiling-Spiegel der DNA-Matrize tragen zum transkriptionalen Bursting bei. Gemäß dem Zwillings-Supercoiling-Domänenmodell kann die Transkription eines Gens die Transkription anderer benachbarter Gene durch ein Supercoiling-Relay beeinflussen. Ein solches Beispiel ist die Aktivierung des leu-500- Promotors. Supercoiling vermittelt nicht nur genspezifische Veränderungen, sondern vermittelt auch groß angelegte Veränderungen der Genexpression. Die topologische Organisation des Nukleoids könnte eine unabhängige Expression von Supercoiling-empfindlichen Genen in verschiedenen topologischen Domänen ermöglichen. Eine genomskalierte Karte des uneingeschränkten Supercoilings zeigte, dass genomische Regionen unterschiedliche stationäre Supercoiling-Dichten aufweisen, was darauf hindeutet, dass das Ausmaß des Supercoilings in einzelnen topologischen Domänen unterschiedlich ist. Als Ergebnis kann eine Änderung des Supercoilings in Abhängigkeit vom Ausmaß des Supercoilings in jeder Domäne zu einer domänenspezifischen Genexpression führen.
Die Wirkung von Supercoiling auf die Genexpression kann durch NAPs vermittelt werden, die das Supercoiling direkt oder indirekt beeinflussen. Die Wirkung von HU auf die Genexpression scheint eine Veränderung des Supercoilings und möglicherweise eine DNA-Organisation höherer Ordnung zu beinhalten. Eine positive Korrelation zwischen DNA-Gyrase-Bindung und Hochregulation der Gene, die durch das Fehlen von HU verursacht wird, legt nahe, dass Veränderungen im Supercoiling für die unterschiedliche Expression verantwortlich sind. Es wurde auch gefunden, dass HU für einen Positionseffekt auf die Genexpression verantwortlich ist, indem Transkriptionseinheiten isoliert werden, indem das transkriptionsinduzierte Supercoiling eingeschränkt wird. Punktmutationen in HUa veränderten das Genexpressionsprofil von E. coli dramatisch und veränderten seine Morphologie , Physiologie und seinen Stoffwechsel . Als Ergebnis war der mutierte Stamm in Säugerzellen stärker invasiv. Dieser dramatische Effekt ging mit einer nukleoiden Verdichtung und einem erhöhten positiven Supercoiling einher. Das mutierte Protein war im Gegensatz zum Wildtyp-Dimer ein Oktamer. Es wickelt DNA auf seiner Oberfläche rechtshändig ein, wodurch positive Supercoils im Gegensatz zu Wildtyp-HU zurückgehalten werden. Diese Studien zeigen, dass Aminosäuresubstitutionen in HU einen dramatischen Einfluss auf die Nukleoidstruktur haben können, was wiederum zu signifikanten phänotypischen Veränderungen führt.
Da sich MukB und HU zu entscheidenden Akteuren bei weitreichenden DNA-Interaktionen entwickelt haben, lohnt es sich, die Wirkung jedes dieser beiden Proteine auf die globale Genexpression zu vergleichen. Obwohl HU die Genexpression durch Modulieren der Supercoiling-Dichte zu kontrollieren scheint, bleibt der genaue molekulare Mechanismus unbekannt und der Einfluss von MukB auf die Genexpression muss noch analysiert werden.
Räumliche Organisation
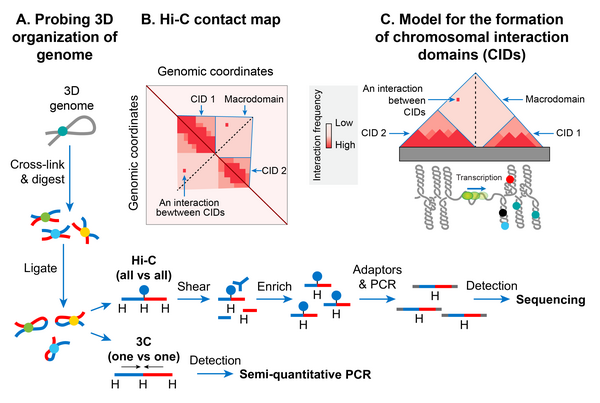
Chromosomale Interaktionsdomänen
In den letzten Jahren hat das Aufkommen einer molekularen Methode namens Chromosomenkonformationserfassung (3C) die Untersuchung einer hochauflösenden räumlichen Organisation von Chromosomen sowohl in Bakterien als auch in Eukaryoten ermöglicht. 3C und seine Version, die mit Deep Sequencing (Hi-C) gekoppelt ist, bestimmen, falls vorhanden, die physische Nähe zwischen zwei beliebigen genomischen Loci im 3D-Raum. Eine hochauflösende Kontaktkarte bakterieller Chromosomen, einschließlich des E. coli- Chromosoms, hat gezeigt, dass ein bakterielles Chromosom in viele stark selbstinteragierende Regionen unterteilt ist, die als chromosomale Interaktionsdomänen (CIDs) bezeichnet werden. CIDs sind äquivalent zu topologisch assoziierenden Domänen (TADs), die in vielen eukaryotischen Chromosomen beobachtet werden, was darauf hindeutet, dass die Bildung von CIDs ein allgemeines Phänomen der Genomorganisation ist. Zwei Merkmale definieren CIDs oder TADs. Erstens interagieren genomische Regionen eines CID physikalisch häufiger miteinander als mit den genomischen Regionen außerhalb dieses CID oder mit denen eines benachbarten CID. Zweitens das Vorhandensein einer Grenze zwischen CIDs, die physikalische Interaktionen zwischen genomischen Regionen zweier benachbarter CIDs verhindert.
Es wurde festgestellt, dass das E. coli- Chromosom in der Wachstumsphase aus 31 CIDs besteht. Die Größe der CIDs reichte von 40 bis ~300 kb. Es scheint, dass eine Supercoiling-Diffusions-Barriere, die für die Auftrennung plektonemischer DNA-Schleifen in topologische Domänen verantwortlich ist, in E. coli und vielen anderen Bakterien als CID-Grenze fungiert . Mit anderen Worten, das Vorhandensein einer Supercoiling-Diffusions-Barriere definiert die Bildung von CIDs. Erkenntnisse aus der Hi-C-Sondierung von Chromosomen in E. coli , Caulobacter crescentus und Bacillus subtilis konvergieren zu einem Modell, das CIDs bilden, weil plektonemische Schleifen zusammen mit DNA-Organisationsaktivitäten von NAPs physikalische Interaktionen zwischen genomischen Loci fördern, und eine CID-Grenze besteht aus eine plektonemfreie Region (PFR), die diese Wechselwirkungen verhindert. Ein PFR wird aufgrund einer hohen Transkriptionsaktivität erzeugt, da die helikale Entwindung von DNA durch aktive Transkription von RNAP plektonemische Supercoils zurückhält. Dadurch wird auch die Dissipation von Supercoils blockiert, wodurch eine Supercoiling-Diffusions-Barriere entsteht. Indirekte Beweise für dieses Modell stammen aus einer Beobachtung, dass CIDs von bakteriellen Chromosomen, einschließlich des E. coli- Chromosoms, an ihren Grenzen hoch transkribierte Gene aufweisen, was auf eine Rolle der Transkription bei der Bildung einer CID-Grenze hinweist. Ein direkterer Beweis kam von einem Befund, dass die Platzierung eines hoch transkribierten Gens an einer Position, an der keine Grenze vorhanden war, eine neue CID-Grenze im C. crescentus- Chromosom erzeugte . Allerdings korrelierten nicht alle CID-Grenzen mit hochtranskribierten Genen im E. coli- Chromosom, was darauf hindeutet, dass auch andere unbekannte Faktoren für die Bildung von CID-Grenzen und Supercoiling-Diffusionsbarrieren verantwortlich sind.
Makrodomänen
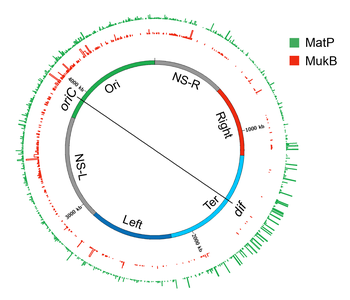
Plektonemische DNA-Schleifen, die als topologische Domänen oder CIDs organisiert sind, scheinen weiter zu verschmelzen, um große räumlich unterschiedliche Domänen zu bilden, die Makrodomänen (MDs) genannt werden. In E. coli wurden MDs zunächst als große Abschnitte des Genoms identifiziert, deren DNA-Marker in Fluoreszenz-in-situ-Hybridisierungsstudien (FISH) zusammen lokalisiert (co-lokalisiert) wurden . Eine große genomische Region (~1-Mb), die den oriC- Locus (Ursprung der Chromosomenreplikation ) abdeckt, war co-lokalisiert und wurde Ori-Makrodomäne genannt. Ebenso war eine große genomische Region (~1-Mb), die die Replikationsterminusregion ( ter ) bedeckte, co-lokalisiert und wurde Ter-Makrodomäne genannt. MDs wurden später basierend darauf identifiziert, wie häufig Paare von Lambda- Att- Stellen, die an verschiedenen entfernten Stellen im Chromosom eingefügt wurden, miteinander rekombinierten. Bei diesem rekombinationsbasierten Verfahren wurde eine MD als eine große genomische Region definiert, deren DNA-Stellen hauptsächlich miteinander rekombinieren können, jedoch nicht mit denen außerhalb dieser MD. Die rekombinationsbasierte Methode bestätigte die in FISH-Studien identifizierten Ori- und Ter-MDs und identifizierte zwei zusätzliche MDs.
Die beiden zusätzlichen MDs wurden durch die zusätzlichen ~1-Mb-Regionen gebildet, die das Ter flankieren und wurden als Links und Rechts bezeichnet. Diese vier MDs (Ori, Ter, Left und Right) machten den größten Teil des Genoms aus, mit Ausnahme von zwei genomischen Regionen, die den Ori flankierten. Diese beiden Regionen (NS-L und NS-R) waren im Vergleich zu einer MD flexibler und unstrukturierter, da DNA-Stellen in ihnen mit DNA-Stellen in MDs auf beiden Seiten rekombiniert waren. Die genetische Position von oriC scheint die Bildung von MDs zu diktieren, da eine Neupositionierung von oriC durch genetische Manipulation zur Reorganisation von MDs führt. Zum Beispiel verhalten sich genomische Regionen, die dem oriC am nächsten sind, unabhängig von der DNA-Sequenz immer als NS, und weiter entfernte Regionen verhalten sich immer wie MDs.
Die Hi-C-Technik bestätigte außerdem eine hierarchische räumliche Organisation von CIDs in Form von Makrodomänen. Mit anderen Worten, CIDs einer Makrodomäne interagierten physikalisch häufiger miteinander als mit CIDs einer benachbarten Makrodomäne oder mit genomischen Loci außerhalb dieser Makrodomäne. Die Hi-C-Daten zeigten, dass sich das E. coli- Chromosom in zwei verschiedene Domänen aufteilte. Die ter umgebende Region bildete eine isolierte Domäne, die mit der zuvor identifizierten Ter MD überlappte. DNA-DNA-Kontakte in dieser Domäne traten nur im Bereich von bis zu ~280 kb auf. Der Rest des Chromosoms bildete eine einzelne Domäne, deren genomische Loci Kontakte im Bereich von >280 kb aufwiesen. Während die meisten Kontakte in dieser Domäne auf eine maximale Entfernung von ~500 kb beschränkt waren, gab es zwei lose Regionen, deren genomische Loci Kontakte in noch größeren Entfernungen (bis zu ~1 Mb) bildeten. Diese losen Regionen entsprachen den zuvor identifizierten flexiblen und weniger strukturierten Regionen (NS). Die Grenzen der isolierten Domäne, die ter und die zwei losen Regionen umfassen, die durch das Hi-C-Verfahren identifiziert wurden, segmentierten das gesamte Chromosom in sechs Regionen, die den vier MDs und zwei NS-Regionen entsprechen, die durch rekombinationsbasierte Assays definiert wurden.
Proteine, die die Bildung von Makrodomänen vorantreiben
MatP
Eine Suche nach Protein(en), die für die Bildung von Makrodomänen verantwortlich sind, führte zur Identifizierung des Makrodomänen-Ter-Proteins (MatP). MATP fast ausschließlich bindet in der Ter MD durch ein 13-bp - Motiv erkennen nannte die macrodomain ter Sequenz ( MATS ). Es gibt 23 matS- Sites in der Ter-Domäne, im Durchschnitt gibt es alle 35 kb eine Site. Ein weiterer Beweis der MatP-Bindung in der Ter-Domäne stammt aus der Fluoreszenz-Bildgebung von MatP. Diskrete MatP-Foci wurden beobachtet, die zusammen mit Ter-Domänen-DNA-Markern lokalisiert waren. Eine starke Anreicherung des ChIP-Seq- Signals in der Ter MD bestätigt auch die bevorzugte Bindung von MatP an diese Domäne.
MatP kondensiert DNA in der Ter-Domäne, da das Fehlen von MatP den Abstand zwischen zwei fluoreszierenden DNA-Markern vergrößerte, die sich in der Ter-Domäne 100 kb voneinander entfernt befinden. Darüber hinaus spielt MatP eine entscheidende Rolle bei der Isolierung der Ter-Domäne vom Rest des Chromosoms. Es fördert DNA-DNA-Kontakte innerhalb der Ter-Domäne, verhindert jedoch Kontakte zwischen den DNA-Loci der Ter-Domäne und denen der flankierenden Regionen. Wie kondensiert MatP DNA und fördert DNA-DNA-Kontakte? Die experimentellen Ergebnisse sind widersprüchlich. MatP kann in vitro einen DNA-Loop zwischen zwei matS- Stellen bilden und seine DNA-Looping-Aktivität hängt von der MatP-Tetramerisierung ab. Die Tetramerisierung erfolgt über Coiled-Coil-Wechselwirkungen zwischen zwei an DNA gebundenen MatP-Molekülen. Ein offensichtliches Modell, das auf In-vitro- Ergebnissen basiert, ist, dass MatP DNA-DNA-Kontakte in vivo fördert, indem es matS- Stellen überbrückt . Obwohl MatP in Hi-C-Studien entfernte Standorte verband, verband es jedoch nicht speziell die matS- Standorte. Darüber hinaus verhielt sich eine MatP-Mutante, die keine Tetramere bilden konnte, wie ein Wildtyp. Diese Ergebnisse sprechen gegen das matS- Brückenmodell für die Ter-Organisation und lassen den Mechanismus der MatP-Wirkung schwer fassbar. Eine Möglichkeit besteht darin, dass MatP sich von seiner primären matS- Bindungsstelle auf nahe gelegene DNA-Segmente ausbreitet und entfernte Stellen über einen Mechanismus überbrückt, der nicht von der Tetramerisierung abhängt.
MukBEF
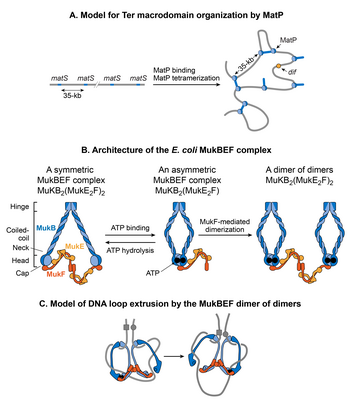
MukB gehört zu einer Familie von ATPasen, die als strukturelle Erhaltung von Chromosomenproteinen (SMCs) bezeichnet werden und an der Chromosomenorganisation höherer Ordnung in Eukaryoten beteiligt sind. Zwei MukB-Monomere assoziieren über eine kontinuierliche antiparallele Coiled-Coil-Wechselwirkung und bilden einen 100 nm langen starren Stab. In der Mitte des Stabes entsteht ein flexibler Scharnierbereich. Aufgrund der Flexibilität des Scharnierbereichs nimmt MukB eine charakteristische V-Form der SMC-Familie an. Die mit MukB assoziierten Nicht-SMC-Untereinheiten sind MukE und MukF. Die Assoziation schließt die V-Bildung, was zu großen ringartigen Strukturen führt. MukE und MukF werden zusammen mit MukB im gleichen Operon in E. coli kodiert . Die Deletion einer der Untereinheiten führt zum gleichen Phänotyp, was darauf hindeutet, dass der MukBEF-Komplex die funktionelle Einheit in vivo ist . Die DNA-Bindungsaktivitäten des Komplexes liegen in der MukB-Untereinheit, während MukE und MukF die MukB-Aktivität modulieren.
Der MukBEF-Komplex wird zusammen mit Topo IV für die Dekatenierung und Neupositionierung neu replizierter oriC s benötigt. Die Rolle von MukBEF ist während der DNA-Replikation nicht eingeschränkt. Es organisiert und kondensiert DNA auch in nicht replizierenden Zellen. Die aktuelle hochauflösende Chromosomen-Konformationskarte des MukB-depletierten E. coli- Stammes zeigt, dass MukB an der Bildung von DNA-DNA-Interaktionen auf dem gesamten Chromosom außer in der Ter-Domäne beteiligt ist. Wie wird MukB daran gehindert, in der Ter-Domäne zu agieren? MatP interagiert physisch mit MukB und verhindert so, dass sich MukB in der Ter-Domäne lokalisiert. Dies zeigt sich in der DNA-Bindung von MatP und MukB in der Ter-Domäne. Die DNA-Bindung von MatP ist in der Ter-Domäne angereichert, während die DNA-Bindung von MukB im Vergleich zum Rest des Genoms reduziert ist. Darüber hinaus verursacht bei einem Stamm, dem MatP bereits fehlt, das Fehlen von MukB eine Verringerung der DNA-Kontakte über das gesamte Chromosom, einschließlich der Ter-Domäne. Dieses Ergebnis stimmt mit der Ansicht überein, dass MatP MukB aus der Ter-Domäne verdrängt.
Wie funktioniert der MukBEF-Komplex, um das E. coli- Chromosom zu organisieren ? Nach derzeitiger Auffassung organisieren SMC-Komplexe Chromosomen, indem sie DNA-Schleifen extrudieren. SMC-Komplexe translozieren entlang der DNA, um Schleifen in einer cis-Weise (auf demselben DNA-Molekül) zu extrudieren, wobei die Größe der Schleifen von der Prozessivität des Komplexes abhängt. SMC-Komplexe aus verschiedenen Organismen unterscheiden sich im Mechanismus der Schlingenextrusion. Einzelmolekül-Fluoreszenzmikroskopie von MukBEF in E. coli legt nahe, dass die minimale funktionelle Einheit in vivo ein Dimer von Dimeren ist. Diese Einheit wird durch die Verbindung zweier ATP-gebundener MukBEF-Komplexe durch MukF-vermittelte Dimerisierung gebildet. MukBEF lokalisiert sich in der Zelle als 1-3 Cluster, die parallel zur Längsachse der Zelle verlängert sind. Jeder Cluster enthält durchschnittlich ~ 8-10 Dimere von Dimeren. Nach dem aktuellen Modell extrudiert der MukBEF DNA-Schleifen auf „felskletternde“ Weise. Ein Dimer der Dimere setzt ein DNA-Segment frei und fängt ein neues DNA-Segment ein, ohne vom Chromosom zu dissoziieren. Neben dem DNA-Looping legt eine Verbindung zwischen negativem Supercoiling und in vivo- MukBEF-Funktion zusammen mit der Fähigkeit der MukB-Untereinheit, negative Supercoils in vitro zu beschränken, nahe, dass MukBEF die DNA organisiert, indem es Supercoils erzeugt.
Rolle von NAPs und naRNAs
NAPs tragen nicht nur zur Chromosomenverdichtung durch Biegen, Verbrücken und Schleifen von DNA in einem kleineren Maßstab (~ 1 kb) bei, sondern nehmen auch an der DNA-Kondensation und -Organisation teil, indem sie weitreichende DNA-DNA-Kontakte fördern. Zwei NAPs, Fis und HU, traten als Schlüsselakteure bei der Förderung von weitreichenden DNA-DNA-Kontakten hervor, die über das gesamte Chromosom hinweg auftreten. Es bleibt zu untersuchen, wie DNA-Organisationsaktivitäten von Fis und HU, die in einem kleineren Maßstab (~1-kb) gut verstanden sind, zur Bildung von weitreichenden DNA-DNA-Wechselwirkungen führen. Nichtsdestotrotz erfordern einige der HU-vermittelten DNA-Wechselwirkungen die Anwesenheit von naRNA4. naRNA4 beteiligt sich auch an der Herstellung von weitreichenden DNA-Kontakten. HU katalysiert einige der Kontakte, nicht alle, was darauf hindeutet, dass RNA mit anderen NAPs an der Bildung von DNA-Kontakten beteiligt ist. HU scheint auch zusammen mit MukB zu wirken, um weitreichende DNA-DNA-Wechselwirkungen zu fördern. Diese Ansicht basiert auf Beobachtungen, dass die Abwesenheit von entweder HU oder MukB eine Verringerung der gleichen DNA-DNA-Kontakte verursacht. Es ist unklar, wie MukB und HU möglicherweise bei der Förderung von DNA-DNA-Interaktionen zusammenwirken. Es ist möglich, dass die beiden Proteine physikalisch interagieren. Während MukBEF große DNA-Schleifen extrudiert, kondensiert und organisiert HU diese Schleifen.
Es gibt Berichte, dass funktionell verwandte Gene von E. coli physikalisch im 3-D-Raum innerhalb des Chromosoms zusammenliegen, obwohl sie genetisch weit voneinander entfernt sind. Die räumliche Nähe funktionell verwandter Gene macht nicht nur die biologischen Funktionen besser unterteilt und effizienter, sondern trägt auch zur Faltung und räumlichen Organisation des Nukleoids bei. Eine kürzlich durchgeführte Studie mit Fluoreszenzmarkern zum Nachweis spezifischer DNA-Loci untersuchte paarweise physikalische Abstände zwischen den sieben rRNA-Operons, die genetisch voneinander getrennt sind (bis zu zwei Millionen bp). Es berichtete, dass sich alle Operons außer rrn C in physischer Nähe befanden. Überraschenderweise zeigten 3C-seq-Studien nicht die physikalische Clusterung von rrn-Operonen , was den Ergebnissen der fluoreszenzbasierten Studie widersprach. Daher sind weitere Untersuchungen erforderlich, um diese widersprüchlichen Beobachtungen aufzulösen. In einem anderen Beispiel bildet GalR ein Interaktionsnetzwerk von GalR-Bindungsstellen, die über das Chromosom verstreut sind. GalR ist ein Transkriptionsregulator des Galaktose-Regulons, der aus Genen besteht, die Enzyme für den Transport und den Stoffwechsel des Zuckers D-Galaktose kodieren. GalR existiert in nur ein bis zwei Herden in Zellen und kann sich selbst zu großen geordneten Strukturen zusammenfügen. Daher scheint es, dass DNA-gebundener GalR multimerisiert, um Ferninteraktionen zu bilden.
Globale Form und Struktur
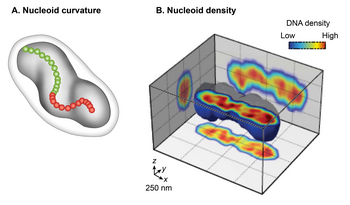
Konventionelle Transmissionselektronenmikroskopie (TEM) chemisch fixierter E. coli- Zellen zeigte das Nukleoid als unregelmäßig geformte Organelle . Die Weitfeld- Fluoreszenzbildgebung von lebenden Nukleoiden in 3D zeigte jedoch eine diskrete, ellipsoide Form. Die Überlagerung eines Phasenkontrastbildes der Zelle und des Fluoreszenzbildes des Nukleoids zeigte nur in der radialen Dimension entlang seiner gesamten Länge des Nukleoids zur Zellperipherie eine enge Aneinanderreihung. Dieser Befund weist auf eine radiale Beschränkung des Nukleoids hin. Eine detaillierte Untersuchung des 3D-Fluoreszenzbildes nach einem Querschnitt senkrecht zu seiner Längsachse zeigte außerdem zwei globale Merkmale des Nukleoids: Krümmung und Längsbereiche mit hoher Dichte. Die Untersuchung der Chiralität der Mittellinie des Nukleoids durch Verbinden der Intensitätszentren jedes Querschnitts zeigte, dass die gesamte Nukleoidform gekrümmt ist. Die Fluoreszenzintensitätsverteilung in den Querschnitten zeigte eine dichte Unterstruktur, bestehend aus gekrümmten Regionen oder Bündeln hoher Dichte im zentralen Kern und Regionen niedriger Dichte an der Peripherie. Eine Implikation der radialen Beschränkung ist, dass sie die gekrümmte Form des Nukleoids bestimmt. Nach einem Modell wird das Nukleoid gezwungen, sich zu biegen, weil es in eine zylindrische E. coli- Zelle eingeschlossen ist, deren Radius kleiner ist als seine biegbare Länge (Persistenzlänge). Dieses Modell wurde durch Beobachtungen gestützt, dass das Entfernen der Zellwand oder die Hemmung der Zellwandsynthese den Radius der Zelle vergrößerte und zu einer gleichzeitigen Zunahme des Helixradius und einer Abnahme der Helixganghöhe im Nukleoid führte.
Nukleoid-Membran-Verbindungen
Eine Expansionskraft aufgrund von DNA-Membran-Verbindungen scheint den Kondensationskräften entgegenzuwirken, um ein optimales Kondensationsniveau des Nukleoids aufrechtzuerhalten. Zellfraktionierungs- und elektronenmikroskopische Studien zeigten zuerst die Möglichkeit von DNA-Membran-Verbindungen. Inzwischen sind mehrere Beispiele für DNA-Membran-Verbindungen bekannt. Transertion ist ein Mechanismus der gleichzeitigen Transkription, Translation und Insertion von naszierenden Membranproteinen, der vorübergehende DNA-Membran-Kontakte bildet. Es wurde gezeigt, dass die Transertion der beiden Membranproteine LacY und TetA die Neupositionierung von chromosomalen Loci in Richtung der Membran verursacht. Ein weiterer Mechanismus von Nukleoid-Membran-Verbindungen besteht in einem direkten Kontakt zwischen membranverankerten Transkriptionsregulatoren und ihren Zielstellen im Chromosom. Ein Beispiel für einen solchen Transkriptionsregulator in E. coli ist CadC. CadC enthält eine periplasmatische sensorische Domäne und eine zytoplasmatische DNA-Bindungsdomäne. Die Wahrnehmung einer sauren Umgebung durch seine periplasmatische sensorische Domäne stimuliert die DNA-Bindungsaktivität von CadC, die dann die Transkription seiner Zielgene aktiviert. Die Membranlokalisation von Genen, die durch einen membranverankerten Transkriptionsregulator reguliert werden, muss noch gezeigt werden. Nichtsdestotrotz wird erwartet, dass die Aktivierung von Zielgenen im Chromosom durch diese Regulatoren zu einem Nukleoid-Membran-Kontakt führt, obwohl es sich um einen dynamischen Kontakt handeln würde. Neben diesen Beispielen ist das Chromosom auch durch Protein-Protein-Interaktion zwischen DNA-gebundenen Proteinen, zB SlmA und MatP, und dem Divisom spezifisch an der Zellmembran verankert . Da Membranprotein-kodierende Gene im gesamten Genom verteilt sind, können dynamische DNA-Membrankontakte durch Transertion als nukleoide Expansionskraft wirken. Diese Expansionskraft würde den Kondensationskräften entgegenwirken, um ein optimales Kondensationsniveau aufrechtzuerhalten. Die Bildung von hochkondensierten Nukleoiden bei Exposition von E. coli- Zellen gegenüber Chloramphenicol, das die Translation blockiert, unterstützt die Expansionskraft transienter DNA-Membran-Kontakte, die durch die Transertion gebildet werden. Die runde Form übermäßig kondensierter Nukleoide nach Chloramphenicol-Behandlung deutet auch auf eine Rolle von durch Transertion vermittelten DNA-Membran-Kontakten bei der Definition der ellipsoiden Form des Nukleoids hin.
Visualisierung
Das Nukleoid kann auf einer elektronenmikroskopischen Aufnahme bei sehr starker Vergrößerung deutlich sichtbar gemacht werden, wobei es zwar anders aussehen kann, aber gegenüber dem Zytosol deutlich sichtbar ist . Manchmal sind sogar DNA- Stränge sichtbar. Durch Färbung mit der Feulgen-Färbung , die spezifisch DNA anfärbt, kann das Nukleoid auch unter einem Lichtmikroskop gesehen werden . Die DNA-interkalierenden Farbstoffe DAPI und Ethidiumbromid werden häufig für die Fluoreszenzmikroskopie von Nukleoiden verwendet. Es hat eine unregelmäßige Form und kommt in prokaryotischen Zellen vor.
DNA-Schäden und -Reparatur
Veränderungen in der Struktur des Nukleoids von Bakterien und Archaeen werden nach Exposition gegenüber DNA-schädigenden Bedingungen beobachtet. Die Nukleoide der Bakterien Bacillus subtilis und Escherichia coli werden beide nach UV-Bestrahlung deutlich kompakter. Die Bildung der kompakten Struktur in E. coli erfordert eine RecA- Aktivierung durch spezifische RecA-DNA-Wechselwirkungen. Das RecA-Protein spielt eine Schlüsselrolle bei der homologen rekombinatorischen Reparatur von DNA-Schäden.
Ähnlich wie bei B. subtilis und E. coli oben verursacht die Exposition des Archaeons Haloferax vulcanii gegenüber DNA-schädigendem Stress eine Verdichtung und Reorganisation des Nukleoids. Die Verdichtung hängt vom Proteinkomplex Mre11-Rad50 ab, der einen frühen Schritt in der homologen rekombinatorischen Reparatur von Doppelstrangbrüchen in der DNA katalysiert. Es wurde vorgeschlagen, dass die Nukleoidverdichtung Teil einer DNA-Schadensreaktion ist, die die Zellwiederherstellung beschleunigt, indem sie DNA-Reparaturproteinen hilft, Ziele zu lokalisieren, und indem sie die Suche nach intakten DNA-Sequenzen während der homologen Rekombination erleichtert.
Siehe auch
Verweise
![]() Dieser Artikel wurde von der folgenden Quelle unter einer CC BY 4.0- Lizenz ( 2019 ) angepasst ( Berichte von Gutachtern ):
Subhash Verma; Zhong Qian; Sankar L. Adhya (Dezember 2019). "Architektur des Nukleoids Escherichia coli" . PLOS-Genetik . 15 (12): e1008456. doi : 10.1371/JOURNAL.PGEN.1008456 . ISSN 1553-7390 . PMC 6907758 . PMID 31830036 . Wikidata Q84825966 .
Dieser Artikel wurde von der folgenden Quelle unter einer CC BY 4.0- Lizenz ( 2019 ) angepasst ( Berichte von Gutachtern ):
Subhash Verma; Zhong Qian; Sankar L. Adhya (Dezember 2019). "Architektur des Nukleoids Escherichia coli" . PLOS-Genetik . 15 (12): e1008456. doi : 10.1371/JOURNAL.PGEN.1008456 . ISSN 1553-7390 . PMC 6907758 . PMID 31830036 . Wikidata Q84825966 .

